|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Location:Home > 実験手法別製品・技術情報 > 2D DIGE(蛍光標識二次元発現差異解析) |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
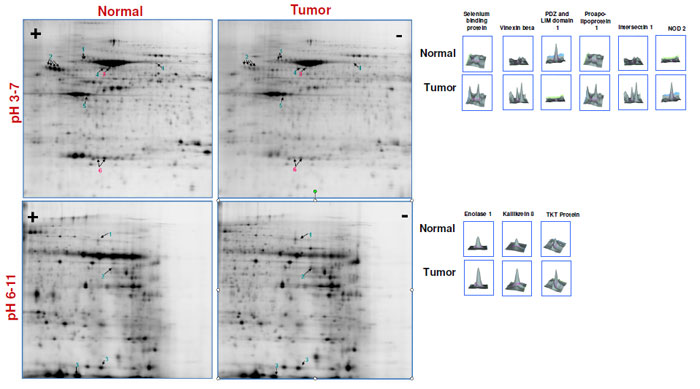
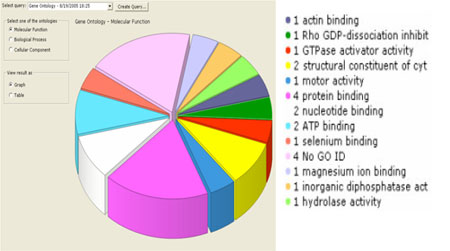
ガン研究のためのバイオマーカー探索D, Haid1 , F. Bahram1, A. Jorsback1, O.Ronn1, A. Wanders2 and H. Bergling. はじめにガンのバイオマーカー探索に代表される臨床研究や創薬研究において、プロテオミクスの技術が活用されてきています。プロテオミクスの技術には蛍光標識二次元ディファレンスゲル電気泳動(2D DIGE)技術と質量分析計(MS)を用いた2D-MS、および液体クロマトグラフィー(LC)と質量分析計(MS)を組み合わせたLC-MSシステムによる解析という2つのアプローチ方法があります。 手法2D-MSのワークフロー健常者と患者の組織をそれぞれ5人ずつ採取し、凍結させた組織をバッファー(8 M urea、2 M thiourea、4 % CHAPS、30 mM Tris、5 mM magnesium acetate)に溶解し、Sample Grinding Kitを用いて破砕しました。pHを8.5に調整した後、2-D Quant Kitを用いてタンパク質濃度を測定しました。測定したサンプルをCyDye™ Fluorで標識しました。5サンプルから等量ずつ混合したものを内部標準として使用しました。一次元目は24 cmのImmobiline™ DryStripを、二次元目は12.5 %のEttan™ DALTゲルを用いて泳動しました。ゲルを固定化しTyphoon™ 9400(後継機種はTyphoon™ FLA 9500)で画像を検出、DeCyder™ 2D ソフトウェアにより解析し、発現量に差のあったスポットをピッキングしてトリプシン消化し、Ettan™ MALDI ToF-Proで同定しました。 MDLCワークフロー健常者と患者のサンプルをアルキル化、トリプシン消化した後にEttan™ MDLCおよびイオントラップ型質量分析計Finnigan™ LTQ(Thermo Electron) で解析しました。1 µgのサンプルをイオン交換カラム(2.1×250 mm SCX、BioBasic、 Thermo Electron)に添加し、塩化アンモニウムでステップ溶出した後に、トラップカラム(0.3×5 mm, Zorbax™, Agilent™)に吸着させ、さらに逆相カラム(0.075×150 mm, Zorbax™, Agilent™)を用いる2次元のクロマトグラフィーで分離しました。逆相分離はA液:0.1 % ギ酸およびB液:84 %アセトニトリル・0.1 % ギ酸を用いて、0~60 % Bを60分のリニアグラジエントで行いました。デュアルカラム(2セットのトラップ・分離カラム)により、LC-MS測定と次回の測定に用いるカラムの平衡化を同時に行いました。MS分析はFull MS/Zoomscan/MS/MSのTriple Play Modeで行いました(on the three most abundant ions)。Dynamic exclusionは30秒で測定しました。タンパク質の同定には、X! Tandem検索エンジンを使用しました。 ※Ettan™ MALDI ToF-Pro、DeCyder™ 2D ソフトウェアは販売終了しておりますが、参考情報として掲載しております。 結果2D-MSのワークフローで得られた結果2D-MSで得られた二つのゲルを比較したところ、発現量が異なるスポット(p-value<0.01)が69個見つかり、39のタンパク質由来であることが分かりました。DeCyder™ EDAの解析で39個のタンパク質を、1)構造タンパク質 2)転写因子、3)シグナル伝達物質。9つのタンパク質の3つのグループに分けることができました。これらのうち9つのタンパク質はすでに他のガンで見つかっているものでした。
図1 ガンおよび正常組織のゲルイメージの比較
図2 DeCyder™ソフトウェアのBVAモジュールのスクリーンショット 表1 同定されたタンパク質の情報
MDLCワークフローで得られた結果MDLC-LTQで得られたMS/MSスペクトルを用いて、Tandem searchアルゴリズムにより、ヒトのデータベース検索を行いました。健常者サンプルでは174個、ガン患者のサンプルでは270個のタンパク質が同定されました(p-value<0.05)。興味深いことに、MDLCワークフローで見つかったバイオマーカー候補は、2D-MSワークフローでは見つかりませんでした。
結論●2D DIGE解析により9個のガンのバイオマーカー候補が見つかり、機能的なグルーピングが行えました。 ●MDLC-LTQからガンに関連する12種類のタンパク質が見つかり、non-differential MDLCがバイオマーカー探索に利用できることがわかりました。 ●2つのワークフローで共通のバイオマーカー候補が見つかっていないことから、今後はゲルベース・LCベースの一方の技術だけではなく、両方の技術を用いた探索の必要性が示唆されました。 お問合せフォーム※日本ポールの他事業部取扱い製品(例: 食品・飲料、半導体、化学/石油/ガス )はこちらより各事業部へお問い合わせください。 お問い合わせありがとうございます。 |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
© 2026 Cytiva