2D-DIGE技術を使った宿主由来タンパク質パターン解析によるプロセス理解の改善
抗体医薬品などでモノクローナル抗体(以後Mabと略します)を製造する際、宿主由来タンパク質(host cell protein, HCP)の混入を防ぐための培養条件検討する際には従来ELISAが用いられてきましたが、2D-DIGE(Two-dimensional Fluorescence Difference Gel Electrophoresis)で、HCPのプロファイルとパターンを比較する手法も注目を浴びています。内部標準を用いてアップストリームとダウンストリームのパターンを定量的に関連付け、分析できます。MAb精製後のサンプル中に残る低レベルのHCPも検出でき、パターンの相違を検出できます。このプロテオミクス的アプローチにより、プロセスの理解を深め、さらにプロセスの最適化、設計による品質の作り込み(Quality by design)に寄与します。
はじめに
モノクローナル抗体などのバイオ医薬品は、遺伝子を改変させた哺乳類細胞系を使用して製造されます。これらの製品の品質を確保し患者への安全性を担保するため、規制当局から厳しく規制されています。したがって、製造プロセス全体を理解することがリスク・アセスメントやプロセス・コントロールにきわめて重要となっています。通常、MAbは1D-SDS電気泳動で純度を測定し、また、残存HCPのレベルは抗体ベースのELISA法で定量されます。2D-DIGEのアプローチでは、これらの手法から得られる情報に加え、サンプル間でのタンパク質のパターンの違い、およびその量の違いが分析可能で、より価値のある情報を入手できます(図1)(1,2)。2D-DIGEは感度が高いため個々のタンパク質はもちろん、微量タンパク質やアイソフォームが検出できます。さらに統計学的に信頼性の高いタンパク質量の差異を分析可能です。
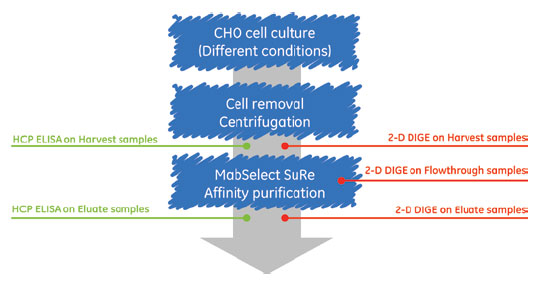
図1. 実験のデザインおよびワークフロー例。MAb発現CHO細胞を種々の条件で培養します。精製前のサンプルと精製後のフラクションを2-D DIGEで解析しました。
特別な抗体を必要とせずそれぞれの工程のHCPsを高感度で検出できます。また、プロセス全体のHCPパターンが得られます。2-D DIGE法では、3種類までのサンプルを同一ゲル内で比較でき、内部標準を使用することで異なるゲル間のサンプルの比較も可能です(3)。サンプルと内部標準は二次元電気泳動の前に、CyDye™ DIGE Fluor minimal dyesによりラベルしました。Cy2、Cy3およびCy5は電荷も分子量も同じなので、それぞれで標識されたタンパク質は、ゲル内を同時に移動します。また、蛍光イメージャーで色素ごとに別々のパターンとして検出できます。
DeCyder™ 2D 7.1 SPNソフトウェアを使用し、内部標準のマップを基準にサンプルのスポットマップを標準化することでゲル間の実験誤差を除き、定量性が高いデータが得られます。DeCyder™ 2D 7.1 SPN ソフトウェアはユーザが指定したスポットでノーマライズするオプション機能も持っています。通常の内部標準が使用できない場合は、異なるスポットパターンを比較するためにCyDye™標識の前に既知のタンパク質を標準サンプルとして加えます。差異が認められたスポットはピッキングしてその後の質量分析計での分析も可能です。
細胞培養時の添加剤がHCPのパターンにどのような影響を及ぼすか、清澄化直後のサンプル(スタート・サンプル)とMabSelect SuRe™(Protein Aベースのクロマトグラフィー担体)でのアフィニティー精製後のサンプルを2D-DIGEを用いて比較しました。UpstreamからDownstreamまでの新し定量方法としてご紹介します。
※DeCyder™ 2Dソフトウェアは販売終了となりましたが、参考情報として掲載しております。
方法
細胞培養
実験概要は図1に示しました。抗体を発現させるチャイニーズ・ハムスター・卵巣(Chinese Hamster Ovary ,CHO)細胞は6 mMグルタミンを含むPower CHO-2 cell culture media を用いて、以下の4条件で100 ml用のフラスコ中で培養しました。
C1 添加剤なしのコントロール
C2 ハイドロライセート、高品質のアミノ酸、ペプチド、ビタミン類および必須栄養素
C3 ハイドロライセートおよび酪酸ナトリウム
C4 リプミン、発現細胞成長因子、トランスフェリン代替物および合成ホルモン
それぞれの条件は3連の実験で行いました。培養終了後、細胞は遠心分離で取り除き、清澄化溶液を分析に使用しました。
力価の定量とELISA
抗体力価の定量には、1 mlのHiTrap™ MabSelect SuRe™のアフィニティークロマトグラフィーカラムを用いました。少量サンプルを中性pHでカラムに添加し、結合物質をpH 3で溶出させました。溶出のピークを280 nmの吸収から得て、スタンダードカーブと比較しました。HCPレベルはELISAを用いて測定しました。
アフィニティー精製
細胞除去後の清澄化溶液中の抗体はHiTrap™ MabSelect SuRe™で精製しました(C1, C2, C4)。PBS中の抗体濃度20 g/Lでサンプルをアプライし、60 mMのクエン酸ナトリウム緩衝液(pH3.5)で溶出しました。溶出液はリン酸ナトリウム溶液で中和しました。細胞除去後の清澄化溶液、素通り画分および溶出画分はVivaSpin 500 Sample Concentrator (MWCO 5000)でDIGE用ローディング緩衝液に置換しました(2-D Protein Extraction Buffer-VにTris緩衝液を最終濃度30 mM(pH 8.5)となるように添加しました)。
2D-DIGE分析
細胞除去後の清澄化溶液のサンプルとプール内部標準液は、Cy2、Cy3およびCy5
CyDye™ DIGE Fluor minimal dyesでラベルしました。50 μgのタンパク質を標識反応に用いました。標識前にプール内部標準を含むすべてのサンプルに、3種類のスパイクサンプルを添加しました。サンプルに添加したスパイクサンプルには、10 ngのトランスフェリン(apo-human plasma)、30 ngのレンチル・レクチンおよび30 ngのミオグロブリン(ウマ心臓由来)が含まれます。
この実験で使用したスパイク・タンパク質はテストされたタンパク質のパネルから選びました。個々のラベルされたタンパク質スポットのシグナル強度と2Dパターンを、抗体のスポット・パターンと比較しました。その結果、HCPや抗体のスポットマップとの重なりはほとんどありませんでした。
標識したサンプルと内部標準を混合し、Immobiline™ DryStrip pH 3-11 NL, 24 cmを用いて一次元目(等電点電気泳動)を行いました。引き続き、DIGEのプレキャスト・ゲルを用いた二次元目を行いました。電気泳動終了後、Typhoon™ 9400を用いてスキャンを取りました(Typhoon™ 9400は販売終了となっております。後継機種のTyphoon™ FLA 9000でも同アプリケーションを行っていただけます)。抗体のスポットはタンパク質濃度が高くシグナル強度が飽和しているため、DeCyder™ソフトウエアを使用した解析時には除外しました。
サンプル間のHCPレベルの差異は、統計学的おいび多変量解析的ツールであるDeCyder™ 2D 7.1 SPNソフトウエアで行いました。条件と生物学的再現性の全体の分散は、Principal Component Analysis (PCA)を用いて測定しました。グループ(条件)間で有意に差異のあるタンパク質スポットの検出には、Differential Expression Analysis methods、average ratioおよびStudent’s t-testを用いました。
結果
細胞除去後の清澄化溶液サンプルの分析
細胞除去後の清澄化溶液は、2D-DIGEの標準プロトコールにて分析を行いました。1689個のタンパク質をC1~4の培養条件から得られたイメージでマッチングを行いました。
タンパク質発現パターンの主成分分析(Principal Component Analysis 、PCA)では、繰り返し実験同士の結果は集合しており、2D-DIGEによる分析の再現性の高さを示します。しかし、異なった条件の実験結果では、タンパク質パターンは全く異なっておりました(図2)。同じグループで近接しているC3、C4とは異なり、C1とC2は全く異なる結果となりました。この結果は、MAbの回収量の結果と近似していました(図3A)。したがって、以降の実験は、C1、C2およびC4にフォーカスして行いました。培養条件による抗体の力価と全HCPのレベルは、培養条件によって異なりましたが、抗体回収量とHCPレベルの間での比例関係はありませんでした(図3)。
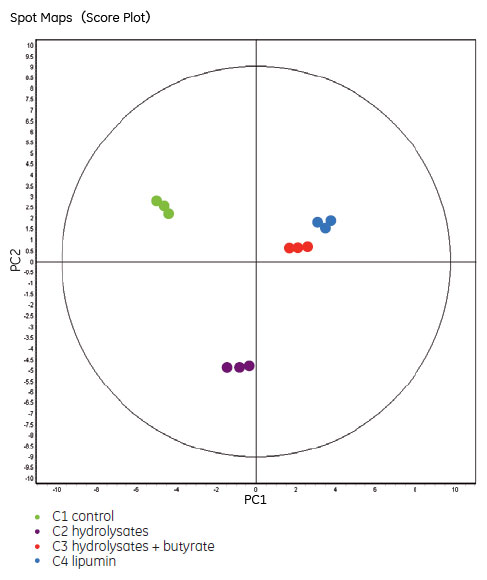
図2. 異なる培養条件におけるクラスタリングを示した主成分分析
それぞれのドットは1つのスポットマップ(1サンプル分のゲルイメージ)を表し、色は培養条件を示しします。
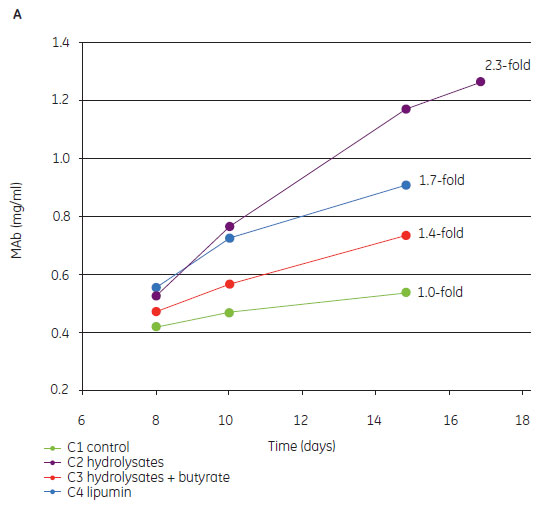
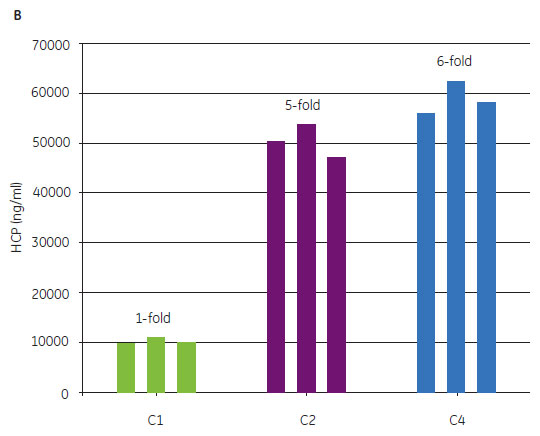
図3. 回収サンプル中の抗体の収量とHCPレベル。 (A) コントロールと比較した最終抗体量(3連の平均値)。培養では約40% Vaibilityの段階で細胞除去を行いました。 (B) 棒グラフの上の数値は、コントロールサンプルのHCPと比較した値を示します。
精製ステップ(Downstream)の分析
2D-DIGEの実験では、それぞれのタンパク質スポットを内部標準で標準化して解析を行いました。シグナル強度についてもサンプル添加量、標識効率およびスキャナーの設定などの差異をなくすために標準化しました。通常の2D-DIGEの実験では、サンプル量が少なくても、条件が違えば大きな差異として検出されるとされています。サンプル間の標準化は、変化の少ないあるいは全く変化のない主要なタンパク質で行います。精製工程のサンプルでは、精製前後のスポットパターンが大きく異なるため、通常のプロテオミクスで使用される標準化は使用できません。精製サンプルを使用した本研究では、代わりに、全サンプルに標識前にスパイクサンプルを添加しました。重なりが少ない3種類のタンパク質で標準化しました。アフィニティー精製における細胞除去した上澄液(スタートサンプル)、素通り画分および溶出画分のサンプルは、プールした内部標準スポットマップ(ゲルイメージ)はDeCyder™ 2D 7.1 SPNで容易にマッチングできました(図4)。また、サンプルは定量的に解析できました。
主要なHCPタンパク質は、予想通りに素通り画分で確認できました。溶出スポットマップでは抗体のアイソフォームが確認できました。図5Aは細胞除去後の上清液、素通り画分および溶出画分の2-Dスポットマップと3-Dビューを示しています。図5Bは溶出画分の1-Dの結果を示します。IgGの軽鎖および重鎖のタンパク質類が2-Dスポットマップ内で横に並んでいることから2-Dの分解能の高さを示しています。
培養条件の違いによる抗体画分に溶出される微量HCPについて、ELISAと2D-DIGEのPCA分析を行いました(図6)。各条件における溶出サンプルのクラスタリングは異なったHCPパターンを示し、精製プロセスで生じる副産物の違いにも影響を与えていることが分かります。溶出グループを発現変動分析(Differential Expression Analysis)で解析し、量的に大きく異なるスポットを見出すことができました。Student’s t-testの値が0.05以下のスポットが96個確認できました。96個のスポットのうち、36個のスポットは抗体およびその副産物に関連していました。60個のスポットがHCPのタンパク質であることが確認されました。HCPに関連したスポットの輪郭線および、副産物の3プロファイルを例として図7に示しました。アウトラインをとったスポットの強度はC1、C2およびC4の条件で有意に異なっていました。この実験では、精製条件が溶出画分のHCPのプロファイルに影響することを示します。
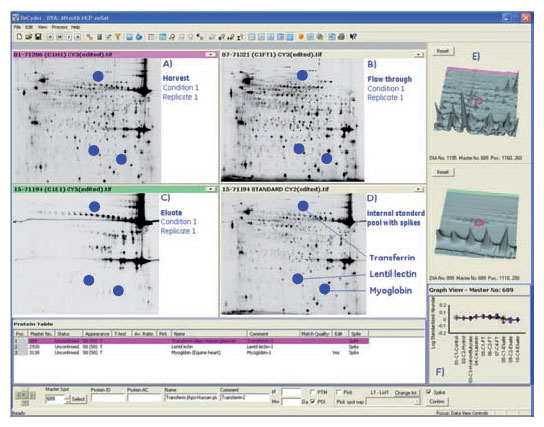
図4. MabSelect SuRe™精製ステップにおけるC1条件のスポットマップ(2連の実験)。(A) 細胞除去後の上澄液(B) 素通り画分、および (C)溶出画分。スポットマップ(D)は印をつけた3種類のスパイクタンパク質と内部標準を示します。3-Dパネル (E) は、スパイクしたトランスへリンと細胞除去後の上澄液(上)と溶出画分(下)を示します。グラフ (F) は、選択したスパイクタンパク質の発現レベル(ログ量)を示します。各データはスパイクタンパク質で標準化しました。
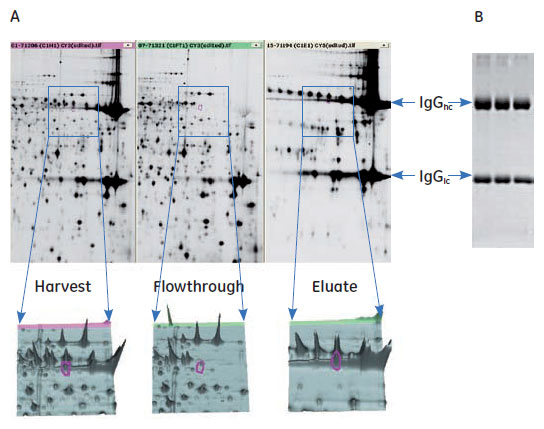
図5. MabSelect SuRe™を用いた精製ステップにおける細胞除去後の上澄液、素通り画分、および溶出画分を分析しました。 (A) スポットマップ(2-Dイメージ)とDeCyder™ 2Dソフトウエアで解析した選択したスポットの3-Dビューを示します。細胞除去後の上澄液、素通り画分、および溶出画分のスポットマップ(50 μgのタンパク質を使用)は非常に異なっていましたが、それぞれのスポットの位置は一致していました。 (B) 同じサンプル5μgを使用した溶出画分の1-D SDS-PAGE分析(Coomassie™で染色)。IgGの軽鎖 (IgGhc) と重鎖(IgGlc) は矢印で示しました。
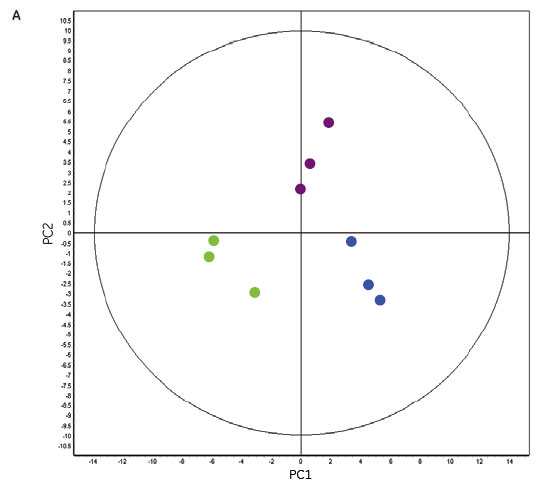
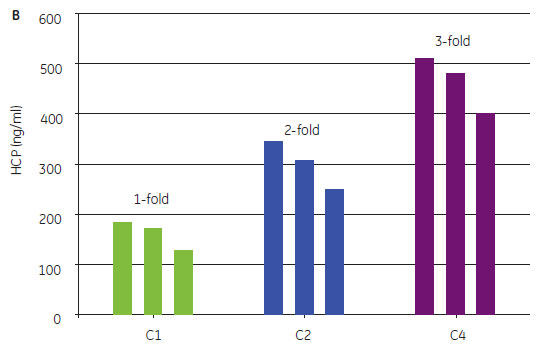
図6. MabSelect SuRe™による精製後の溶出画分を分析しました。 (A) PCAは溶出サンプルは条件によってクラスタライズされることを示ています。 (B) ELISAで分析した溶出画分のHCP量は126~508 ng/ml (18~62 ppm) でした。
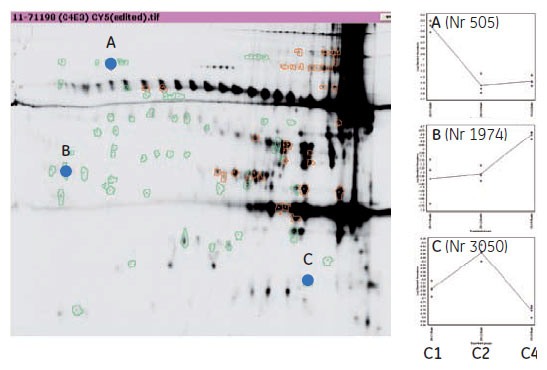
図7. ゲルイメージは溶出されてくるHCPのアウトラインを示しています。緑色に囲われたスポットはHCP関連、オレンジ色に囲われたスポットは抗体関連のスポットです。右側のグラフA、BおよびCは条件C1、C2およびC4における3種類のスポットの発現プロファイルを示します。
まとめ
2-D DIGEは抗体に依存せずに高い精度と感度でひとつひとつのHCPの変化から全体のパターンの変化までを解析できます。1-D SDS-PAGEよりも、精度と感度ともに高く解析できます。そのため、精製の段階で除去が困難なHCPも検出できます。培養および精製全体におけるHCPパターンの変化をモニターすることで、プロセスを優位にコントロールできます。2-D DIGEを用いたHCPの検出はELISAを補完し、クオリティー・オブ・デザイン(Quality of Design)の優れたツールとなりえます。
文献
- Unlü, M. et al. Difference gel electrophoresis: a single gel method for detecting changes in protein extracts. Electrophoresis 11, 2071-2077 (1997).
- Minden, J. S. et al. Difference gel electrophoresis. Electrophoresis, Review, Jun 30 S1:S156-61 (2009).
- Alban, A. et al. A novel experimental design for comparative two dimensional gel analysis: Two Dimensional difference gel electrophoresis incorporating a pooled internal standard. Proteomics 3, 36-44 (2003).
- Jin, M. et al. Profiling of host cell proteins by two dimensional difference gel electrophoresis (2D-DIGE): Implications for downstream process development. Biotechnology and Bioengineering 105, 306-316 (2010).










