|
|||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|
|||||||||||||||||||||||||||||
|
Location:Home > 実験手法別製品・技術情報 > タンパク質サンプル調製・前処理 |
|||||||||||||||||||||||||||||
第5章
|
|||||||||||||||||||||||||||||
| Volume of beads (µl) | 5 | 4 | 3 | 2 | 1 | No beads |
|---|---|---|---|---|---|---|
| Amount of IgG loaded (µg) | 100 | 80 | 60 | 40 | 20 | 20 |
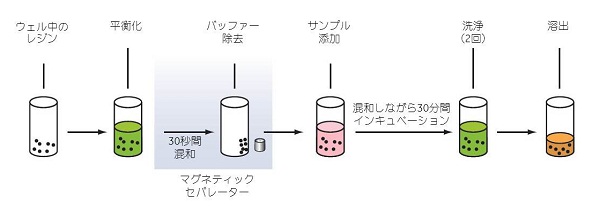
図5.7 結合実験のステップ
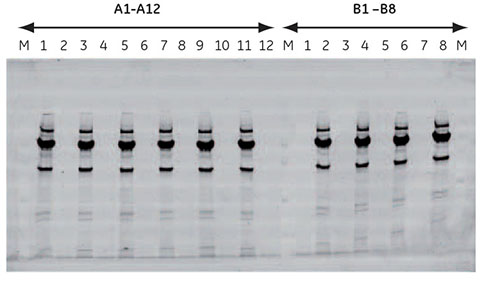
各ウェルについて280 nmで吸光度を測定し、溶出したタンパク質の量を測定しました(図5.8)。96ウェルプレートの8つの列で同等のパターンが見られました。ビーズ容量が等しいウェルで同等量のタンパク質が溶出され、ビーズを入れなかったウェルではタンパク質は測定されませんでした。コンタミネーションは認められませんでした。
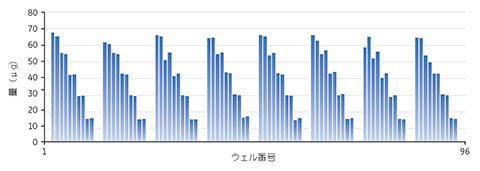
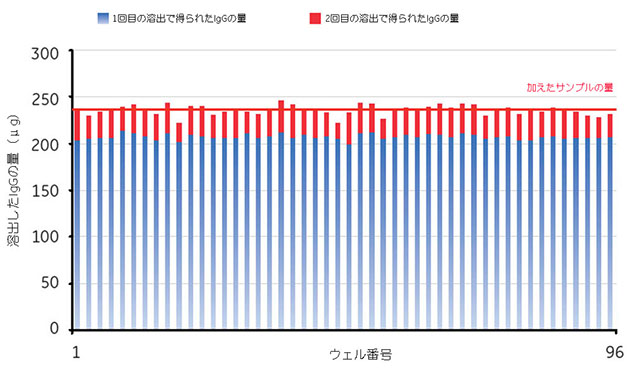
図5.8 A280で測定した各ウェルの溶出IgG量
His Mag Sepharose™ Niを用いた少量のヒスチジンタグGFPサンプルの精製
この実験では、GFP-(His)6を大腸菌溶解液に添加しました。His Mag Sepharose™ Niの使用量によってタグタンパク質の量を調整しました(表5.2参照)。各容量のビーズについて16回反復して実験を行いました。この実験には96ウェルプレートを使用しました。結合バッファーおよび溶出バッファーの組成は次の通りです。結合バッファー=20 mMリン酸ナトリウム、500 mM NaCl、20 mMイミダゾール、pH 7.4、溶出バッファー=20 mMリン酸ナトリウム、500 mM NaCl、500 mMイミダゾール、pH 7.4、溶出したタンパク質を280 nmにおける吸光度で測定し(図5.9A)、SDS-PAGEで泳動しました(図5.9B)。磁気ビーズおよびサンプルの容量にかかわらず、同じ純度が得られました。GFP-(His)6の収量の測定間変動はきわめて小さく、RSD(相対標準偏差)は2%~5%でした。
表5.2 実験に使用したビーズの容量とGFP-(His)6の量
| Volume of beads (µl) | 5 | 4 | 3 | 2.5 | 2 | 1 |
|---|---|---|---|---|---|---|
| Amount of GFP-(His)6 loaded (µg) | 94 | 75 | 56 | 47 | 38 | 19 |
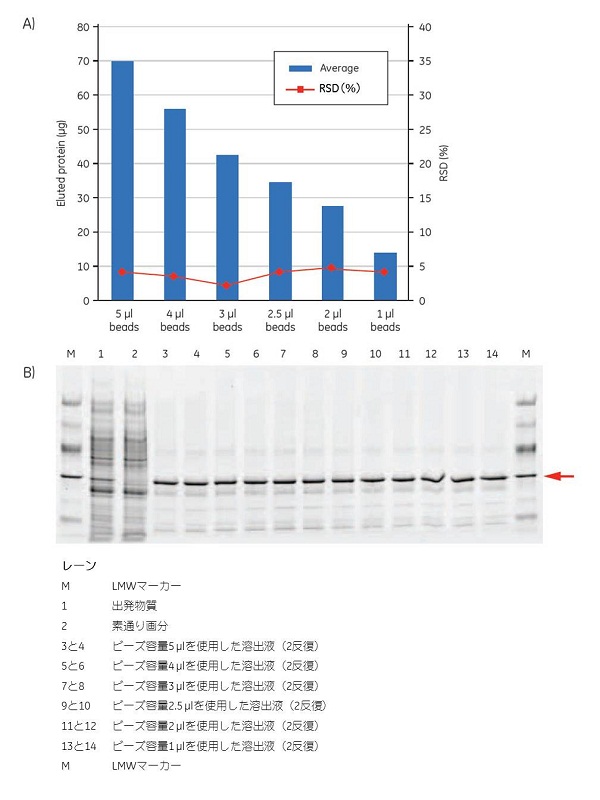
図5.9 (A)A280測定値による溶出タンパク質量の平均値とそのRSD(相対標準偏差)(%)。(B)出発サンプル、素通り画分および6通りのビーズ容量を用いたときの溶出画分(ビーズ容量につき2回反復)を泳動させたSDSポリアクリルアミドゲル。SDS-PAGE画像中の矢印はGFP-(His)6のMrを示します。
スクリーニングアプリケーション
MultiTrap™およびMag Sepharose™フォーマットは、タグ組換えタンパク質のハイスループットタンパク質生産ワークフローにおいてスクリーニングを行う場合に特に適しています。このようなワークフローでは (3、4)、多数のタンパク質について特徴付けを行う必要があり、これを達成するために一般に各タンパク質について複数のコンストラクトのスクリーニングと最適化を行います。このプロセスを効率的に扱うためには、並行作業が不可欠です。組換えタンパク質や抗体の発現スクリーニングと条件スクリーニングに適用される一般的なプロトコールを図5.10に示します。
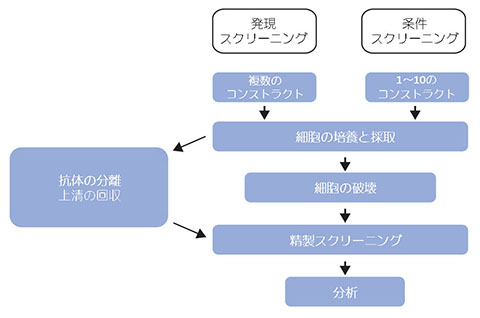
図5.10 発現スクリーニングと条件スクリーニングのワークフロー
GSTタグおよびヒスチジンタグタンパク質の濃縮に使用するMultiTrap™製品では、各ウェルに最大600 μlのサンプルをアプライでき、マイクログラムから数ミリグラムの濃縮タンパク質が得られます。GST MultiTrap™およびHis MultiTrap™製品では、サンプルを清澄化せずに直接アプライできることから、必要な調製時間が短縮されます。抗体生産の条件に関するスクリーニングについても同じ手法を使用でき(Protein A HP MultiTrap™/Protein G HP MultiTrap™プレートを使用)、この場合、多数のハイブリドーマのスクリーニングを平行して行うことが可能です。
タグを持つ、またはタグを持たない組換えタンパク質の精製のスケールアップはこのハンドブックでは扱いませんが、多くの場合、小スケールで使用した条件と方法をスケールアップしたフォーマットに簡単に移行できます。
※販売終了品を含みますが参考情報として掲載しております。
タンパク質サンプル調製ハンドブック目次 |5章 References |略号と用語、記号解説
お問合せフォーム
※日本ポールの他事業部取扱い製品(例: 食品・飲料、半導体、化学/石油/ガス )はこちらより各事業部へお問い合わせください。
お問い合わせありがとうございます。
後ほど担当者よりご連絡させていただきます。
© 2026 Cytiva