Evaluation software上では、データの解析時にkineticsかaffinityか、ボタン一つ押すだけで解析できてしまいます。ではKinetics解析とaffinity解析で得られたKDは同じなのでしょうか?Affinity解析はkinetics解析と異なりリガンド量はあまり気にしなくて良い…と言われているのはなぜでしょうか?
色々と面白いaffinity解析のあれこれについて解説したいと思います。
Kinetics解析との違い、どういう時にAffinity解析するか?
Kinetics解析はkaやkdを算出したい時、Affinity解析は平衡値に達した時
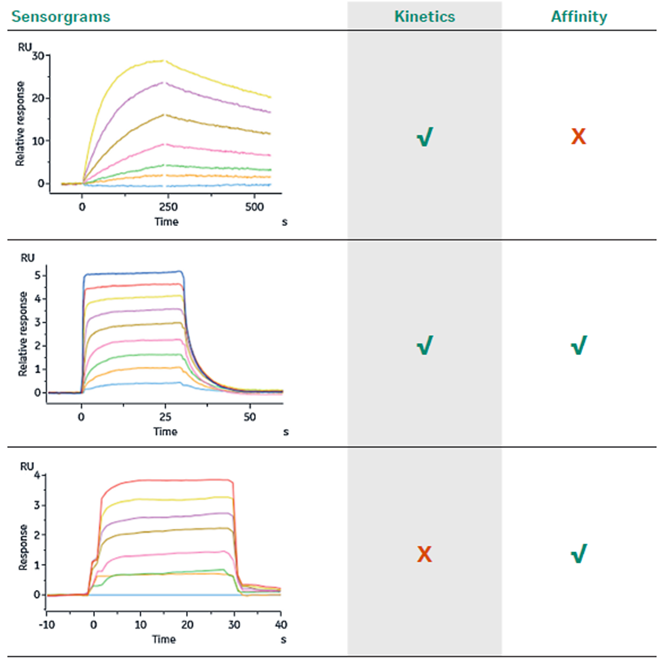
Figure 1

Figure 1
Kinetics解析を実施するのは ka や kd といった反応速度論的パラメータを算出したい場合です。その際はその相互作用における結合様式が明確であり、センサーグラムが緩やかにカーブしており、フィットさせるモデル式が用意されている必要があります。一方でAffinity解析を実施するのは解離速度定数KDを算出したい場合です。比較的解離が速く、箱形のセンサーグラムを描く場合に用いられ、Figure 1のように各濃度のレスポンスが平衡値に達している必要があります(= 「添加中に」 各濃度のレスポンスが頭打ちになる必要があります)。平衡値に達していないデータを解析することはできません。
平衡状態に達しない場合は基本的に添加時間を伸ばして対応するしかありません(低濃度側で見られがち)。ただし解離がある程度遅い相互作用の場合はいくら添加時間を伸ばしても平衡状態に達さないケースも出てきます(後述)。そういった相互作用ではaffinity解析を正しく行うことができません。
計算式から学ぶ、Affinity解析の意味
Affinity解析は1:1 binding
Affinity解析、という名前から分かるようにこの解析方法はAffinityを算出する解析方法です。Affinityとは1:1 bindingの親和力であり、KDで表記されます。Kinetics解析では KD = kd / ka という形で表現されますが、解離が非常に速い相互作用の場合は ka も kd もそもそも算出できません。そもそも、KD は以下のように定義されています。
A(Analyteとします)と B(Ligandとします)の相互作用において、ABをAとBの複合体とし、以下のような相互作用があるとします。
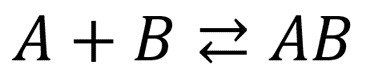
AB複合体の結合速度係数をka、解離速度定数をkdと表記すればAB複合体の濃度の変化率は以下のように記述できます。
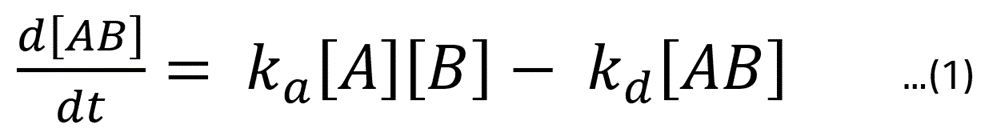
平衡状態において(1)式の左辺は0に近似できますから、式(1)は変形すれば式(2)になります。これはKDの定義です。
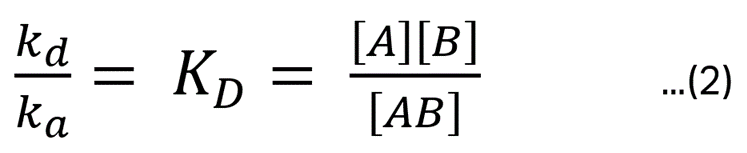
アナライトを特定の濃度で添加したときの平衡状態におけるAB複合体の濃度を [AB] = x とすると、式(2)のKDの定義は式(3)のように記述できます。
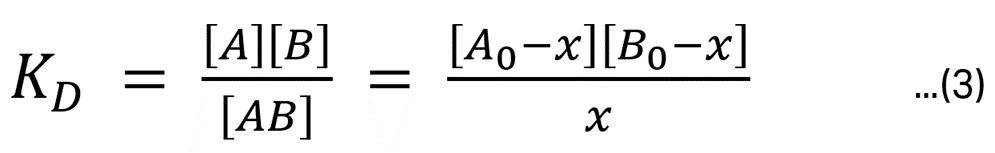
ここで A0, B0はそれぞれアナライトとリガンドの初期濃度とします。Biacore™は流路系の測定系ですからアナライトは添加中は常に一定の濃度に保たれます。バイアルなどに用意したアナライトの添加濃度をCと置けば、[A0 – x] = C と記述できます。また Bfを複合体を形成していないフリーなリガンドの濃度とすれば、 [B0 -x] = Bf と記述することができますから、式(1)のKDは以下のように記述し直すことができます。
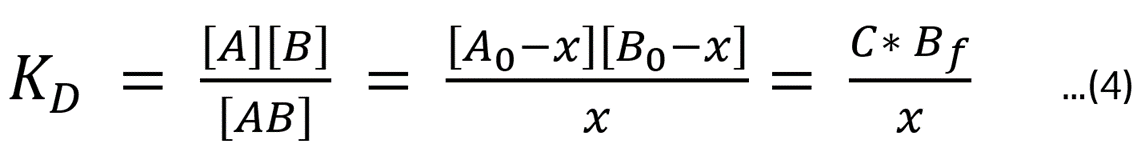
ではここでアナライトの濃度Cについて、KDという濃度で添加したとします。すると式(4)は x = Bf と記述されます。つまりアナライトがKDという濃度で添加されたのであれば、平衡状態においてフリーなリガンドの数と複合体ABを形成している数が等しいことを示しています(Figure 2)。別の言い方をすれば、センサーチップ上に固定化されたリガンド全てが複合体を取るときレスポンスの半分のレスポンスを与えるときのアナライト濃度がKDという濃度です(Figure 3)。なお、Reqとはそれぞれの濃度における平衡値を指します。
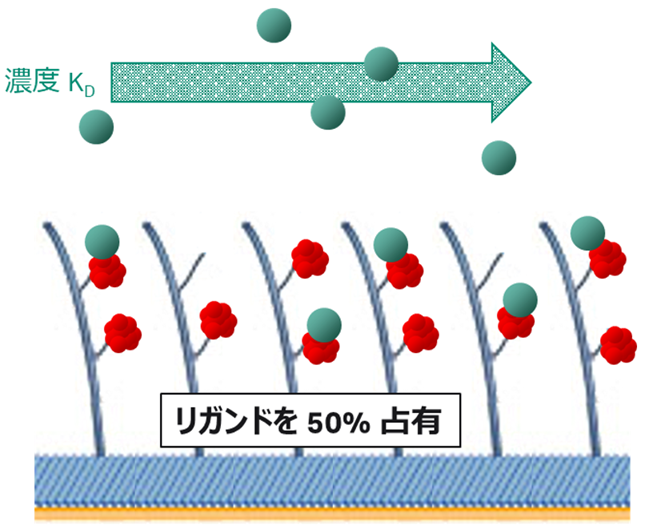
Figure 2:アナライト濃度KDで添加した時の平衡状態の様子
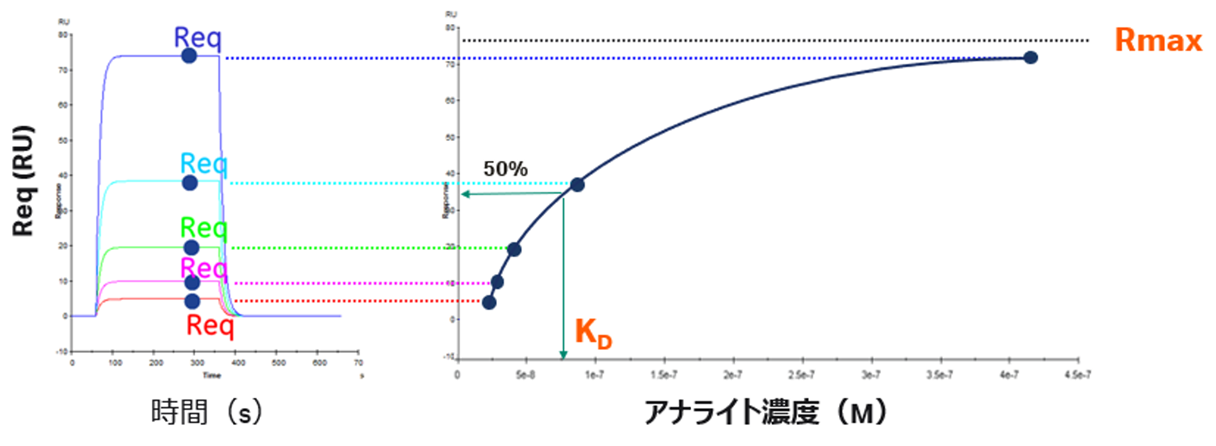
Figure 3
ではここでアナライト濃度Cを10* KD あるいは 1/10* KD にしてみたらどうなるか考えてみたいと思います。この時式(4)はそれぞれ x = 10*Bf あるいは10*x = Bf と記述されますので、それぞれFigure 4のようになります。
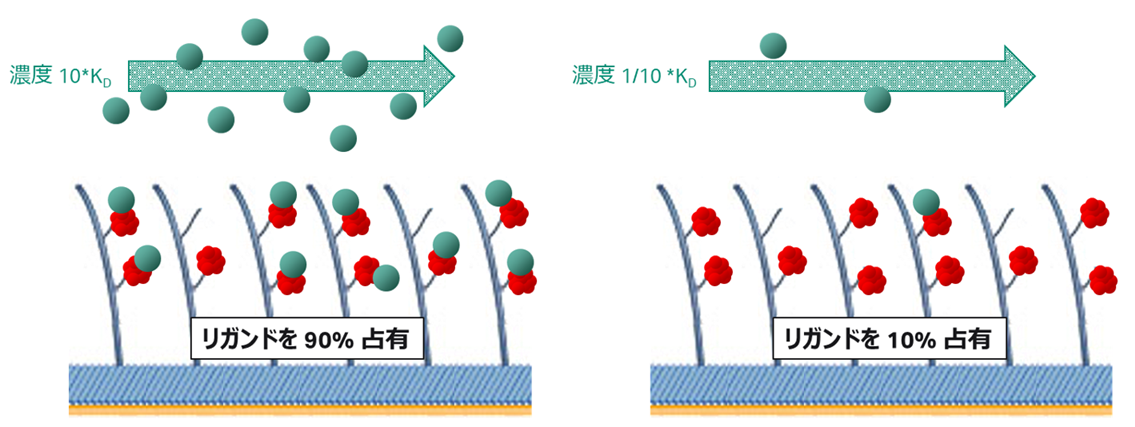
Figure 4
もう少し詳細に、式(4)をレスポンスに換算してみます。濃度の単位Mからレスポンスの単位RUに変換する因子Gを用い、さらに得られる複合体の形成により得られるレスポンスをRとすれば、
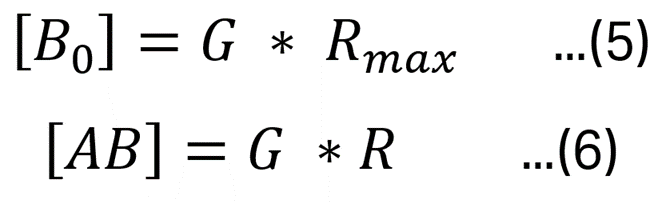
と記述できるので、
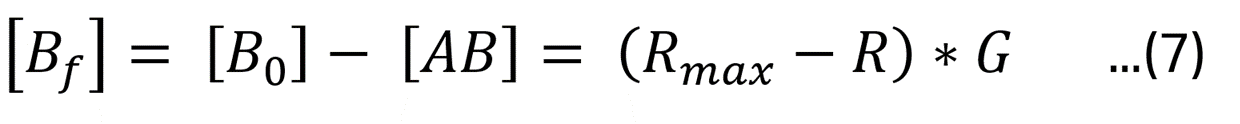
なお平衡状態においてRはReq と記述できるので、式(7)は式(8)のように記述できます。
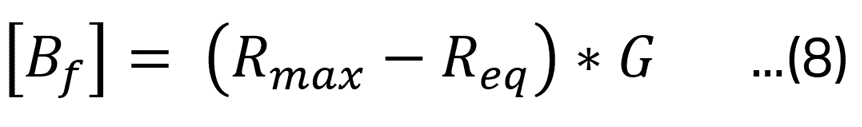
すると式(4)は
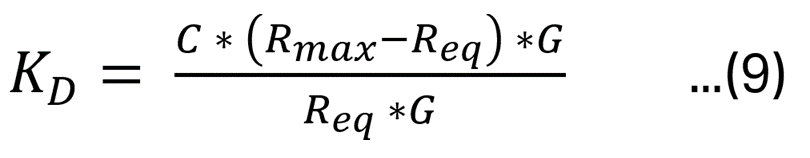
式(9)をReq について解けば、式(10)が導かれます。
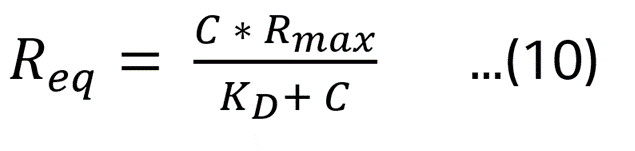
特に、C = KD の時、式(10)は 2 * Req = Rmax となり、平衡値はリガンドの量に依存していないことが分かります(Figure 5 および式(5)-(10))。
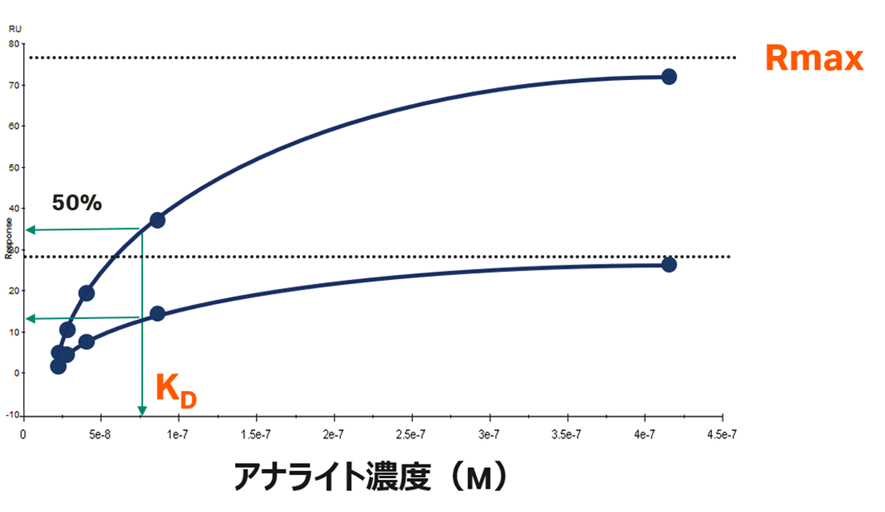
Figure 5:リガンドの量でReq自体は変わるがKDは変わらない
1:1 bindingではない相互作用をaffinity解析していいの?(見かけのKDとは?)
前章のように、affinity解析は1:1 bidingを想定した親和力の算出でした。一方で、この解析方法はとりあえず各濃度で添加したアナライトによるレスポンスが平衡状態にさえなっていれば、1:1 bindingではない相互作用についても解析することが可能であり、KD という数値は算出できてしまいます(見かけの KD などと呼ばれたりします)。本来はこうしたKD は科学的な意味はないのですが、何らかの数値を出したい、という方が利用することがあります。
シンプルな1:1 bindingではないので KD の値はリガンドの量により変動してしまいます。同じリガンド量であれば相対的な比較は可能と考えられますが、基本的にこの値だけで親和力を議論することは難しいと思われます。
解離が遅い相互作用の場合は平衡値に達しにくい
例えばFigure 6ではka一定、kdがそれぞれ異なる相互作用においてアナライト濃度KDで添加した場合です。計算するとkdが精々1e-2より大きいくらいでないとaffinity解析はできないことが分かります(Table 1)。
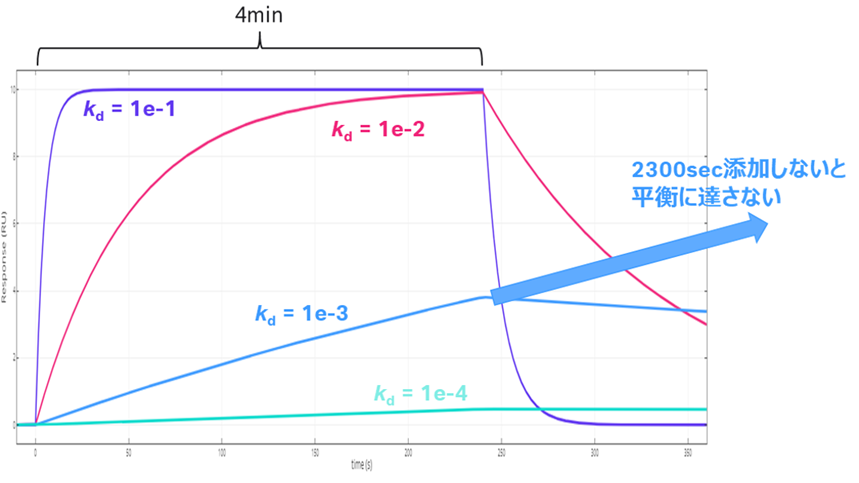
Figure 6:ka=1e6(一定)、添加時間240sec、添加濃度KDにおける平衡状態
| ka | kd | KD(nM) | アナライト添加濃度 | |||
|---|---|---|---|---|---|---|
| 100*KD | 10*KD | KD | 0.1*KD | |||
| 1e6 | 1e-1 | 100 | 0.5 | 4 | 23 | 42 |
| 1e6 | 1e-2 | 10 | 5 | 42 | 230 | 419 |
| 1e6 | 1e-3 | 1 | 46 | 419 | 2303 | 4187 |
| 1e6 | 1e-4 | 0.1 | 456 | 4187 | 23026 | 41865 |
Table 1:平衡値の99%に達するまでに必要な時間(sec)
Affinity解析における添加濃度条件
濃度条件が適切でないとどうなるか?
教科書的にはしばしば「affinity解析では8点以上の濃度データが必要で、最高濃度は少なくとも2*KD以上の濃度が必要」と記載されています。実際、解析画面でも2*KDの濃度が取得できていないとKDを示す線が赤色に表示され、高濃度のデータが不足していることが警告されます。
ここで少し実験的に、Carbonic AnhydraseⅡに対してFurosemideを添加してaffinity解析を行ったデータに対して色々と条件を変えて解析してみたいと思います。
まずは完全な形のデータから見てみます。
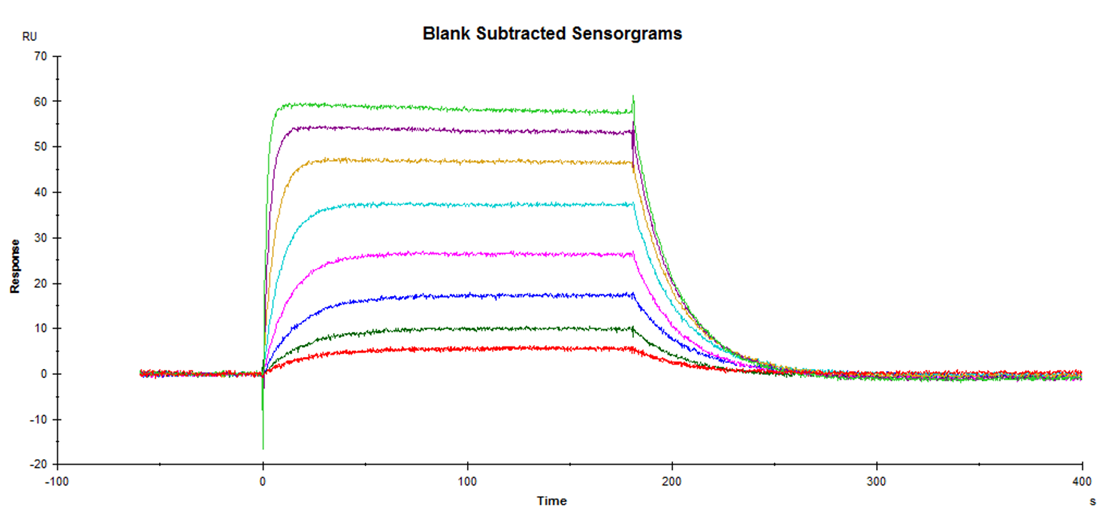
Figure 7
最高濃度10 μMから2倍希釈系列で8点、最低濃度は79 nMです。全ての濃度サンプルは平衡状態に達しており、理想的なaffinity解析が可能です。
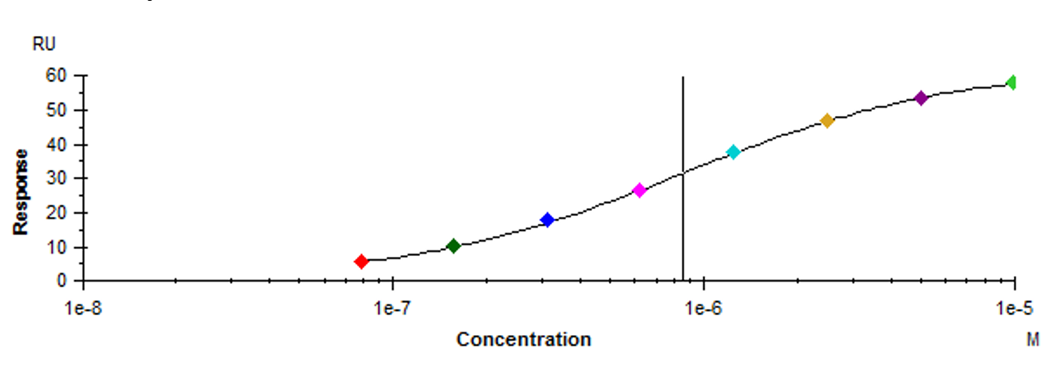
Figure 8
これをsteady state affinity解析するとFigure 8のようになりました(KD = 854 nM)。この図ではKDは1e-6よりももう少し小さいくらいに算出されることが見て取れます。
※見慣れないかもしれませんがFigure 8では濃度が対数表示されております。これには低濃度側が見やすくなる、希釈公比が同じならx軸に対して等間隔でデータが並ぶ、中点がKDとなるといった特徴があります。
ではここから高濃度側のデータを一点ずつ削除して再解析してみます。
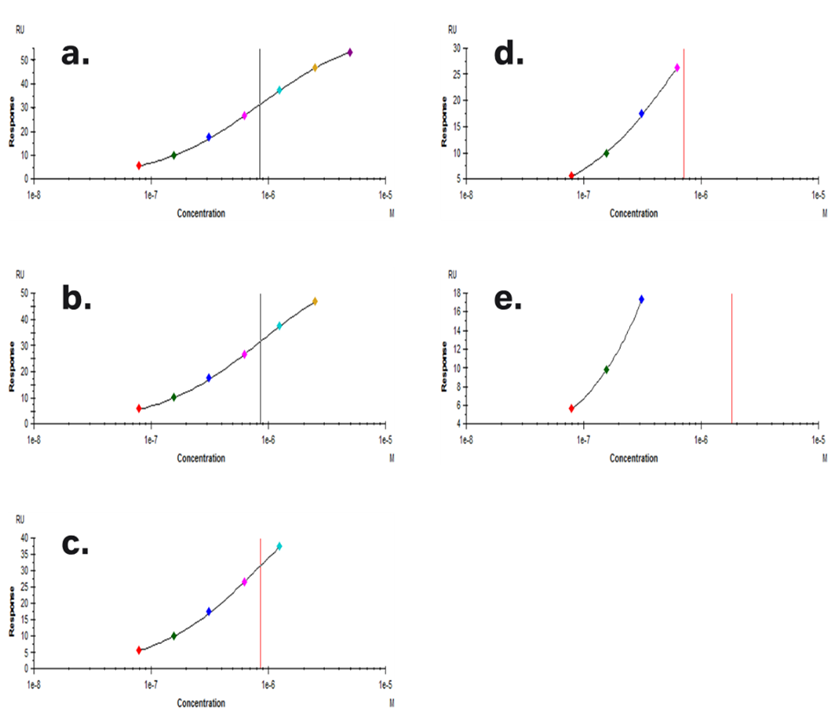
Figure 9
続いて、低濃度側のデータを一点ずつ削除して再解析してみます。
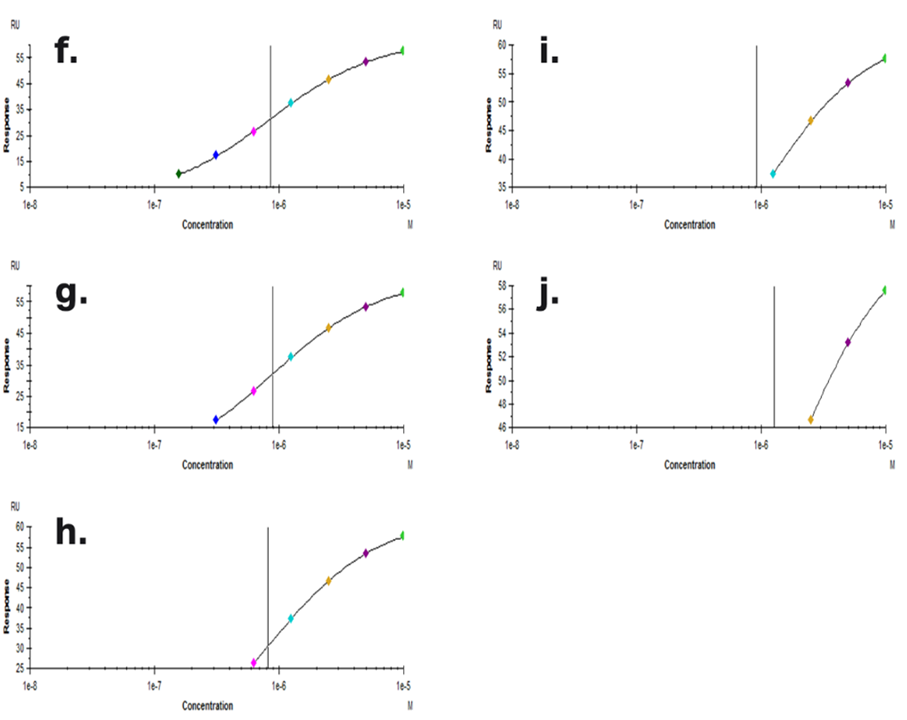
Figure 10
それぞれの算出値を表にまとめると以下の通りです。
| Original | a. | b. | c. | d. | e. | f. | g. | h. | i. | j. | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| KD(nM) | 854 | 846 | 854 | 853 | 712 | 1837 | 854 | 886 | 818 | 921 | 1268 |
| Rmax | 62.1 | 62.9 | 62.1 | 62.1 | 56.6 | 112.2 | 62.0 | 61.4 | 63.4 | 59.5 | 49.0 |
| offset | 0.3 | 0.3 | 0.3 | 0.3 | -0.2 | 0.9 | 0.3 | 1.2 | -1.2 | 3.0 | 14.1 |
| Req point number | 8 | 7 | 6 | 5 | 4 | 3 | 7 | 6 | 5 | 4 | 3 |
| Analyte conc. range(X*KD) | 11.71~0.093 | 5.85~0.093 | 2.93~0.093 | 1.46~0.093 | 0.73~0.093 | 0.37~0.093 | 11.71~0.18 | 11.71~0.37 | 11.71~0.73 | 11.71~1.46 | 11.71~2.93 |
想定の通りですが、高濃度のデータが欠けるとRmaxがずれ始め、offsetもずれ、KDもずれることが分かります(a-e)。dの解析結果は受け入れるか微妙なところですが、最高濃度が1.46*KDでありながらも、cは問題なさそうです。大体KDを示す縦線がフィッティングカーブを横切るくらいになっていればまずまずの解析ができそうなことが分かります。
これは低濃度側のデータを削除した場合も同様で、やはりiの解析結果が微妙なところになり、結局こちらも縦線がフィッティングカーブを横切るかどうかくらいが目安になりそうです。
Affinity解析においては高濃度側のデータが不足することはよくあります。よくあるのは、低分子化合物をアナライトとしていて、溶解性の悪いサンプルは高濃度側で沈殿したり凝集してしまって単分散しなくなり、得られたデータが正しくない場合です。このようなときは高濃度側のデータをexcludeして解析することはしばしば実施されます。ある意味これは気付きやすい課題です。一方で低濃度側のデータに関してはあまり注目して見ていないことがあるのではないでしょうか。改めてFigure 7をご覧いただくと、Furosemideは低濃度のデータもきれいに平衡状態に達していることが分かります。しかし、もう少し解離が遅いAzosulfamideなどでは明らかに低濃度側で平衡状態に達していないことが分かります(Figure 11)。こういった相互作用の場合はピンク色のデータよりも高い濃度のサンプルくらいしか解析に用いることができません。解離が遅い相互作用の場合は低濃度側のデータが不足しがちなので注意して見てください。
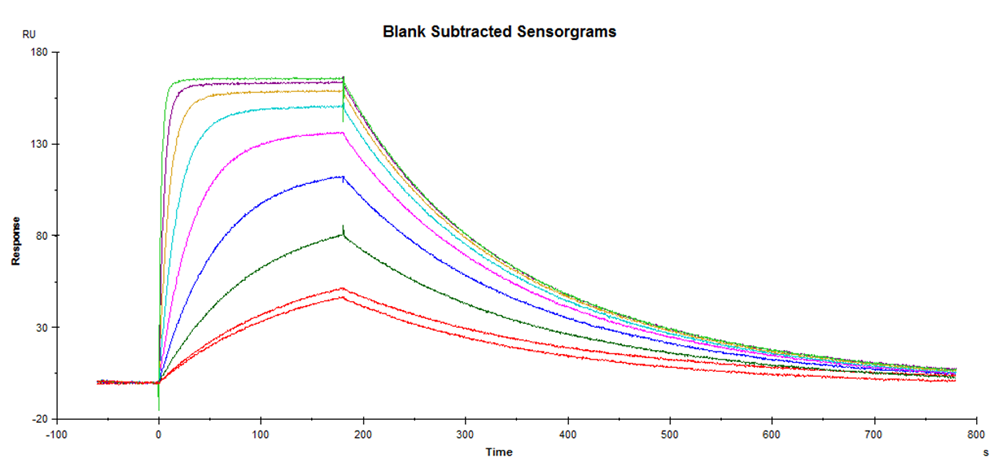
Figure 11
以上、affinity解析のあれこれについて解説いたしました。