Q フィッティングがかかりません!こんな感じです。どうすればいいですか?
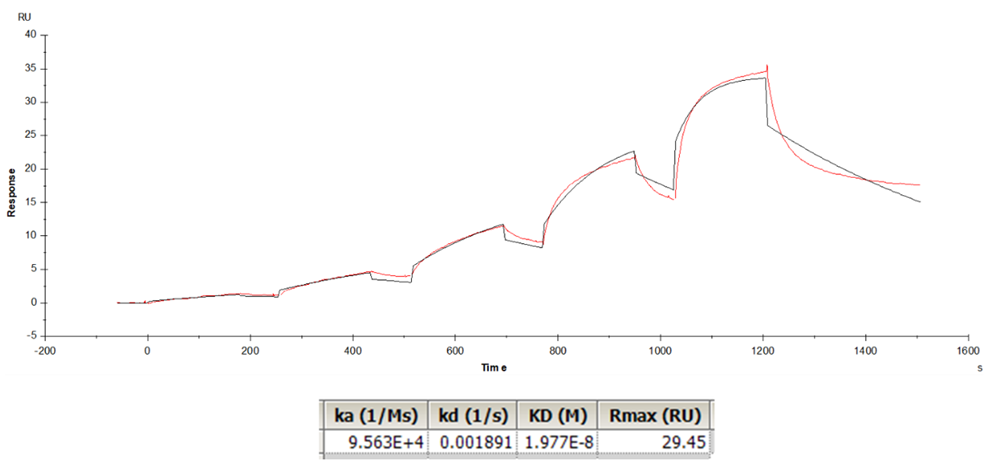
図1.1:1 binding modelでのフィッティング結果
A 着眼点:センサーグラム形状を読み取りましょう。
結構きれいなセンサーグラムに見えるのになのになぜフィッティングしないの、と思われた方もいるかもしれません。でも、Biacore™のデータを見慣れてくると、あぁ、1:1の相互作用ではないな、ということがすぐに分かるようになります。
ポイントは、解離相のカーブの形状が“二相性(あるいは多相性)”になっていないか、というところです。最後の1200secから始まるセンサーグラムの解離相が二相性になっていることが読み取れますか?これは見慣れていないと判断しづらいかもしれませんが、図2のような見方をすると分かりやすいかもしれません。このようなセンサーグラム形状は少なくとも1:1の相互作用では絶対に起こりえない形状です。
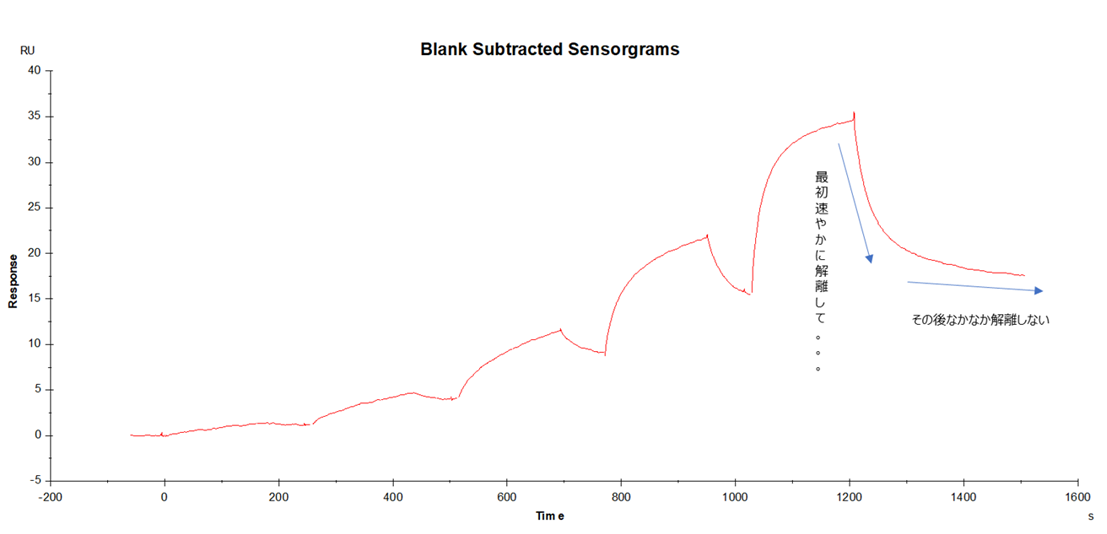
図2.二相性のセンサーグラムの読み方
それではこのケースで何が起こっているのかを説明したいと思います。
実はこのケースでは、アナライト分子が2量体だったものです。結論から言いますと、Biacore™の測定系の構築において、サンプルに2量体や抗体IgGなど1分子に2つの等価の結合部位が存在する分子はリガンド(固定化)側にすることを優先します。アナライト側にしてしまいますと、このようにリガンドに対して“1本の手”で結合している成分と“2本の手”で結合している成分が混在し、このように二相性のセンサーグラム形状になり、その結果1:1 binding model でのフィッティングが不良になります(つまり、そもそも1:1 binding modelが適用不可のセットアップである、ということです)
となると、次の打ち手としては、以下が考えられます。
- Bivalent analyte modelを適用する
- 2価の分子側をリガンド(固定化)側にして再測定する
①の場合は前号のBivalent Analyte modelでも述べましたが、数値の信頼性を担保するのが 1:1 binding modelに比べて難しくなります。本記事はBiacore™初心者向けのものですので、以下にも簡単にその理由を書きたいと思います。
Bivalent Analyte modelにおける“2本目の手“の結合は(1本目の時の結合と異なり)センサーチップ上にすでに存在するアナライトがセンサーチップ表面構造のデキストランなどが密集する環境で固定化されている分子同士で起こる現象です。ですので、モデル式に記述されている2段階目の数式項が実際の挙動を適切に反映しているかはわかりません(極論を言うと全てのモデル式は大なり小なりそのような側面もありますが、特にこの2段階目の挙動の実際のところはわかりえないところと言えます)。
なので、出来るだけ固定化量を低くして、立体的なリガンド間の距離を長くして、2段階目の結合があまり多くは起こらない環境で測定するというやり方もあるのですが、もし可能なら②の方法で測定する方が信頼性の高い数値を得やすくなることが多いと思います。
ここで、①の方法、つまりBivalent Analyte modelを用いてフィッティングすると、図3のように比較的きれいにフィッティングがかかります。ですが、上述した通り2段階目を記述する数式項は現実の挙動を正しく反映しているのかかなり不明なものですので、いろいろ条件を振って同じような解(ka1、kd1)が出るかなど、数値の信頼性を検証したほうが良いかもしれません。
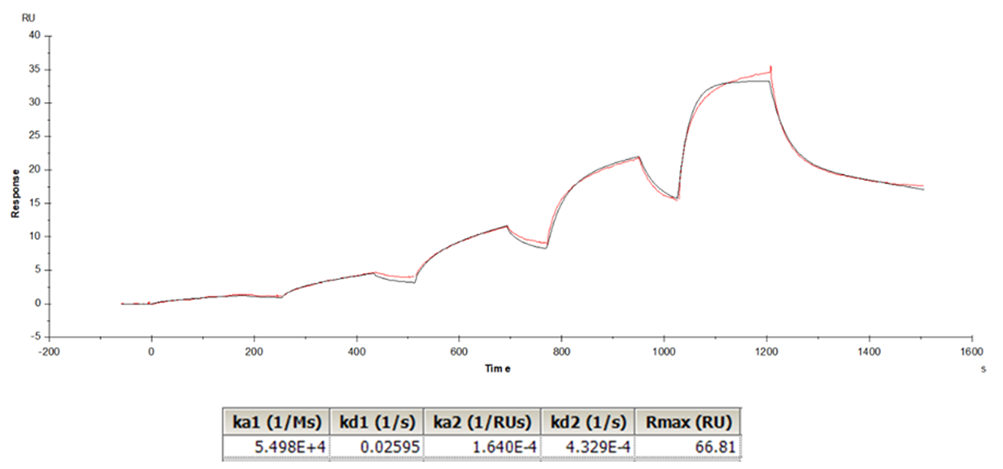
図3.Bivalent Analyte modelを用いたフィッティング
そのような検証作業まですることを考えると、②の2量体分子側をリガンドにするのが、フィッティング解の信頼性の面において一般的には優先順位の高い方法と言えます。
ここで試しにBiacore™シミュレーションで得ることができるWebツール“Biacore™ Simul8”(マニュアルはこちらから)を利用して、図3で得られたka1, kd1を用いて、この2量体分子をリガンドとした場合を想定したセンサーグラムを図4に示します。
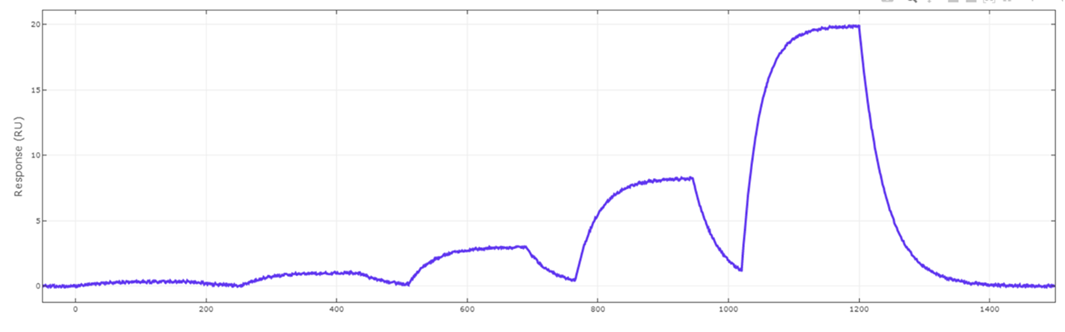
図4.2量体側をリガンドとした場合のセンサーグラム(シミュレーション)
うーん、二相性の無いほれぼれするようなきれいな1:1 bindingのセンサーグラム形状ですね!