抗体精製をマスターしよう (8)
モノクローナルIgGの高純度精製
 第7回「モノクローナルIgGの精製プロトコール」では、モノクローナルIgG精製の一般プロトコールについてご紹介しました。Protein G/A を用いたアフィニティークロマトグラフィー精製は特異性の高い精製方法ですが、精製IgG中に微量のタンパク質が混在することがあります。このような場合は、イオン交換、疎水性相互作用、ゲルろ過クロマトグラフィーのステップを追加して純度を高めます。
第7回「モノクローナルIgGの精製プロトコール」では、モノクローナルIgG精製の一般プロトコールについてご紹介しました。Protein G/A を用いたアフィニティークロマトグラフィー精製は特異性の高い精製方法ですが、精製IgG中に微量のタンパク質が混在することがあります。このような場合は、イオン交換、疎水性相互作用、ゲルろ過クロマトグラフィーのステップを追加して純度を高めます。
また、失活しやすい不安定な抗体の場合には、精製条件の至適化をすることで純度や安定性を高めることができます。
今回は、4つの精製例から、効率的に高純度のモノクローナルIgGを精製するためのスキームをご紹介します。
マウスモノクローナルIgGの精製条件至適化と自動二段階精製
Protein A やProtein G を利用したアフィニティークロマトグラフィーによるモノクローナルIgGの精製は便利な手法ですが、動物種やサブクラスの種類によって抗体の親和性が異なるため、担体への結合、解離条件を至適化する必要があります。
ここではIgG1をHiTrap™ rProtein Aで精製するときの条件検討例と溶出画分をゲルろ過クロマトグラフィーでさらに精製した例について紹介します。ここではIgG1はProtein A カラムへの結合が弱いので、結合バッファーの条件を至適化する必要がありました。条件検討にはÄKTA™FPLCの自動スカウティング機能を利用しました。
精製操作
-
モノクローナルIgG1を含む細胞培養上清(90 ml)
-
-
HiTrap™ rProtein A による精製(回収)
-
結合バッファーの最適塩濃度の決定
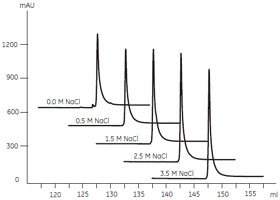
- サンプル:モノクローナルIgG1を含む細胞培養上清 90 ml
- カラム:HiTrap™ rProtein A, 1ml
- 結合バッファー:100 mM リン酸ナトリウム, 0~3.5 M NaCl, pH7.4
- 溶出バッファー:100 mM クエン酸ナトリウム, pH3.0
- 流速:1 ml/min
- システム:ÄKTA™FPLC
図1 HiTrap™ rProtein Aと抗体の結合に最適な塩濃度の自動スカウティング
まず、HiTrap™ rProtein A カラムへの最適結合条件の検討を行いました。ÄKTA™FPLC で0 ~ 3.5 M の範囲で様々なNaCl 濃度の結合バッファーを自動調製し、結合条件の自動スカウティングを行った結果、NaCl 濃度が2.5 M の場合に抗体とrProtein A カラムとの結合が最適(ピーク面積が最大)となることがわかりました。
溶出バッファーの最適pHの決定
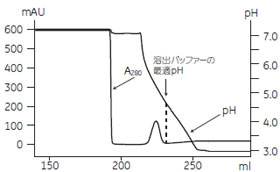
- サンプル:モノクローナルIgG1を含む細胞培養上清 90 ml
- カラム:HiTrap™ rProtein A, 1ml
- 結合バッファー:100 mM リン酸ナトリウム, 100 mM クエン酸ナトリウム, 2.5 M NaCl, pH7.4
- 溶出バッファー:100 mM リン酸ナトリウム, 100 mM クエン酸ナトリウム, pHグラジエント 7.4~3.0
- 流速:1 ml/min
- システム:ÄKTA™FPLC
図2 抗体溶出の最適pH条件の検討
続いて、溶出液のpH条件検討を行いました。一般にProtein A カラムからの溶出にはpH 3.0 の酸性溶液を使用します。不安定な抗体の場合には失活する恐れがあるため、よりマイルドな溶出条件を検討します。pH 7.4 ~ 3.0 のpH グラジエント溶出を行った結果、IgG1 はpH 4.5 で溶出されることが分かりました。
-
ゲルろ過クロマトグラフィーによる最終精製
-
モノクローナルIgGの全自動精製
ÄKTA™FPLCを使用し、HiTrap™ rProtein A カラムから溶出されるIgG ピークをSuperloop™内に回収して、ゲルろ過カラムに自動添加する全自動2 段階精製を行った結果、約1.2 mg のモノクローナルIgG1を精製できました。全自動で精製することにより精製時間の短縮、大量サンプル処理、モノクローナル抗体のルーチン精製が可能になりました。
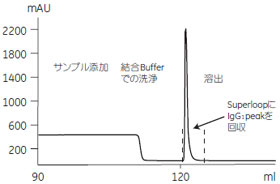
- サンプル:モノクローナルIgG1を含む細胞培養上清 90 ml
- カラム:HiTrap™ rProtein A, 1ml
- 結合バッファー:100 mM リン酸ナトリウム, 2.5 M NaCl, pH7.4
- 溶出バッファー:100 mM クエン酸ナトリウム, pH4.5
- 流速:1 ml/min
- システム:ÄKTA™FPLC
図3 HiTrap™ rProtein Aと抗体の結合に最適な塩濃度の自動スカウティング
上記の検討から至適化した条件により、アフィニティー精製を行いました。
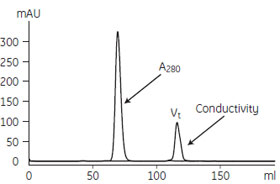
図4 ゲルろ過クロマトグラフィーによる最終精製
続いて、溶出液のpH条件検討を行いました。一般にProtein A カラムからの溶出にはpH 3.0 の酸性溶液を使用します。不安定な抗体の場合には失活する恐れがあるため、よりマイルドな溶出条件を検討します。pH 7.4 ~ 3.0 のpH グラジエント溶出を行った結果、IgG1 はpH 4.5 で溶出されることが分かりました。
-
SDS-PAGEによる純度確認
-
SDS-PAGEによる純度確認
ÄKTA™FPLCを使用し、HiTrap™ rProtein A カラムから溶出されるIgG ピークをSuperloop™内に回収して、ゲルろ過カラムに自動添加する全自動2 段階精製を行った結果、約1.2 mg のモノクローナルIgG1を精製できました。全自動で精製することにより精製時間の短縮、大量サンプル処理、モノクローナル抗体のルーチン精製が可能になりました。
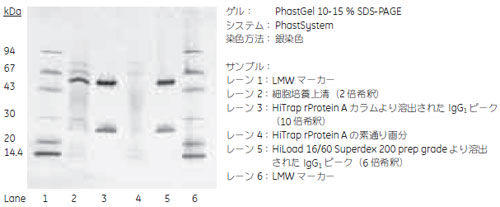
図5 SDS-PAGEによる純度確認
残存するTransferrin やBSA の除去
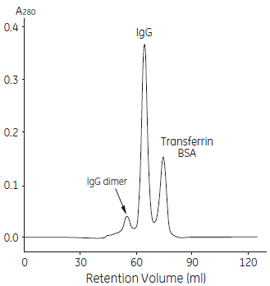
Protein A やProtein G から溶出したIgG 画分にTransferrin(76 kDa)やAlbumin(67 kDa)が含まれる場合は、分子量の違いを利用してゲルろ過クロマトグラフィーで分離することができます。ここでは、ヒトIgG、Albumin、Transferrin の混合物をHiLoad™ 16/60 Superdex™ 200pg で分離した例を示します。
図7 ヒトIgG, Albumin, Transferrin の混合物の分離例
- サンプル:ヒトIgG( 3.6 mg), BSA( 2 mg), transferrin(0.75 mg)を 1 ml に調製
- HiLoad™ 16/60 Superdex™ 200pg
- バッファーA: 20 mM リン酸ナトリウム, 0.15 M NaCl, pH 7.0
- 流速: 0.5 ml/min(15 cm/h)
- 検出:280 nm
サンプルを添加後、120 ml の溶出バッファーで溶出を行いました。
分子量の違いを利用して、ヒトIgG(モノマー)を分離することができました。
脱離したアフィニティーリガンドの除去
通常、Protein A やProtein G カラムからのアフィニティーリガンドの漏出はきわめて微量で、ほとんどの目的にはそのまま使用しても問題ありません。しかし、医薬品などの製造においてはアフィニティーリガンドの混入は深刻な問題であり、微量であっても最終精製画分から除去することが求められています。
アフィニティーリガンドが溶出画分に混入している場合、精製後の確認をSDS-PAGE で行うと、Protein G は30 kDa(実際の分子量は17 kDa ですがSDS-PAGE では構造が広がり移動度が小さくなります)、Protein A は42 kDa バンドとして現れます。さらに高純度が要求される場合には、ゲルろ過クロマトグラフィーやイオン交換クロマトグラフィーによってこれらのリガンドを効果的に除去することができます。
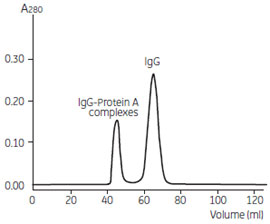
ゲルろ過クロマトグラフィーによるアフィニティーリガンドの除去
図8 ゲルろ過クロマトグラフィーによるrProtein A の除去例
- サンプル:4.8 ml のマウスIgG2a (14 mg), rProtein A(0.36 mg)
- HiLoad™ 16/60 Superdex™ 200 prep grade(120 ml)
- バッファー: 20 mM リン酸ナトリウム, 0.15 M NaCl, pH 7.0
- 流速: 2.0 ml/min(60 cm/h)
- 検出:280 nm
ゲルろ過クロマトグラフィーによるrProtein A の除去例です。
Protein A やProtein G は最大2 分子のIgG と結合します。このようにIgG が過剰の場合、混入するProtein A やProtein G とIgG の複合体は、IgG 単量体の2 倍以上の分子量となるため、ゲルろ過クロマトグラフィーで容易に分離することができます。
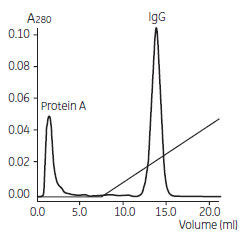
イオン交換クロマトグラフィーによるアフィニティーリガンドの除去
図9 陽イオン交換クロマトグラフィーによるrProtein A の除去例
- サンプル:マウスIgG2b(0.61 mg), rProtein A(1.8 mg)
- HiTrap™ SP 1 ml
- 結合バッファー: 20 mM クエン酸ナトリウム, pH 5.2
- 溶出バッファー: 結合バッファー+ 1.0 M NaCl, pH 5.2
- 溶出条件: 0 ~ 45 %B in 15 CV(カラム体積)
- 流速:1.9 ml/min(296 cm/h)
- 検出:280 nm
陽イオンクロマトグラフィーによるrProtein A の除去例です。
あらかじめ調製したIgG-rProtein A 複合体を、解離条件(rProtein A カラムからの溶出条件)でHiTrap™ SP カラムに添加しました。pH 5.2 では、rProtein A はカラムに結合しないため、効果的な分離が可能です。目的のIgG が陽イオン交換体に対してrProtein A よりも強く結合する場合に適用できます。
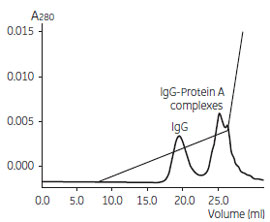
図10 陰イオン交換クロマトグラフィーによるrProtein A の除去例
- サンプル:マウスIgG2a (0.15 mg), rProtein A(0.009 mg)
- HiTrap™ Q(1 ml)
- 結合バッファー: 20 mM Tris-HCl, pH 8.5
- 溶出バッファー: 結合バッファー+ 1 M NaCl, pH 8.5
- 溶出条件: 0 ~ 25 %B in 20 CV(カラム体積)
- 流速:1.9 ml/min(296 cm/h)
- 検出:280 nm
陰イオン交換カラムでもProtein A を除去可能な場合があります。漏出したProtein A やIgGProtein A 複合体はIgG よりも僅かに強く陰イオン交換体担体に結合することを利用します。しかし、多くの場合、Protein A とIgG-Protein A 複合体のピークは明確に分離しないため、精製各フラクションのrProtein A をEIA などにより定量する必要があります。
より純度の高いモノクローナルIgG の精製法についてご紹介しました。ゲルろ過クロマトグラフィーやイオン交換クロマトグラフィーを駆使することで、不純物を除くことができます。ご紹介した例を参考に、高純度精製にチャレンジしてみてください。