Parameter Settings利用したことありますか?
もしかしたら「それなに?」、という方もいるかもしれません。確かにちゃんと理解しないで使うと間違った値を導きかねないところもありますが、うまく使うことにより測定系のセットアップや状況に合わせてより信頼性の高い数値を得ることができます。
まずParameter Settingsとはどこのことかを図1にお示しします。Parameters…というボタンなどを押すと出てくる、この表のことです。
ここでのパラメーター設定項目は以下になります。
- Fit:global か local か constant か
- Initial Value:初期値の設定
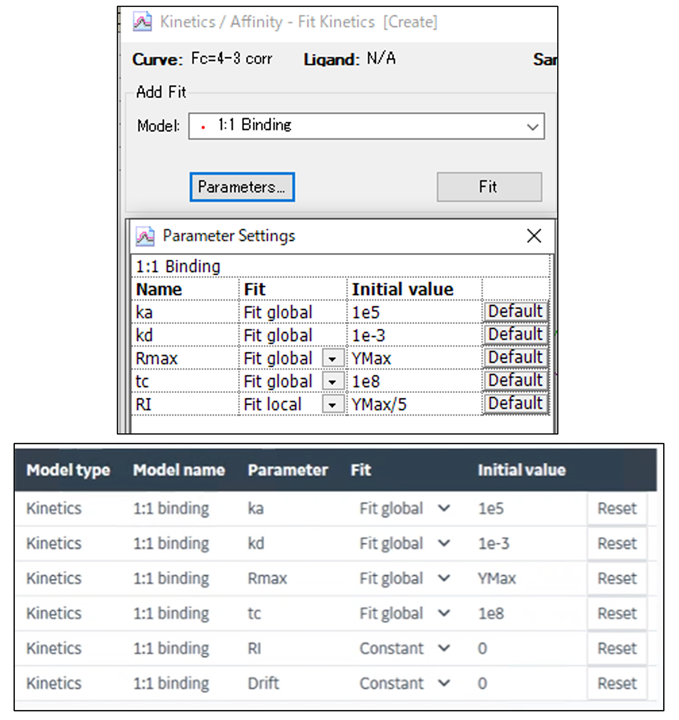
図1:Parameter settings (上段:Biacore™ T200, S200, X100のEvaluation softwareのイメージ、下段:Biacore™ Insight Evaluation Softwareのイメージ)
1.の3つの選択肢ですが、まずconstantは指定の(変数ではない)数値に固定する、ということですので分かりやすいと思います。残りの2つを理解するためには、まずBiacore™のkinetics解析では異なる濃度のアナライトを添加した複数(典型的には5濃度以上)のデータセット(センサーグラム)を用いて解析している、ということを思い出してください。実は1濃度のデータでもフィッティングはできるわけですが5濃度の添加をしているわけです(その理由は過去記事の“5濃度+0濃度、n=2”をご参照下さい)。
そのため、例えば5濃度のセンサーグラムそれぞれからka、kdなどの数値を個別に算出することもできるのですが、実際に皆さんが解析結果をご覧になるときは一組のka、kdを見ていると思います。
上記の前者のような解析をLocal fittingといい、後者のような解析をGlobal fittingと言います。そしてそれは、各変数でどちらにするかを決めて解析することが可能です(一部ソフトウエアで制限をかけている部分はありますが)。
では、それらの選択肢の選び方をパラメーターごとにご紹介いたします。
① ka、kd
- 常にglobalにして解析します。ソフトウエアでglobalから変更できないようにしています。(Biacore™ Insight Evaluation Software (以下Insight)では変更可能ですが、globalを利用)
- ka, kdは、濃度に依存しない、その相互作用固有の数値です。Localでフィッティングしたほうがより実カーブにフィットする(Chi2が小さい)ことが多いですが、globalで解析することでより頑健なka,kdが算出されます。
② Rmax
- globalにするのが基本ですが、以下の条件で変更することがあります。
- local:アナライト各濃度の添加サイクル毎にリガンドの固定化量や結合活性率が異なることが確認、強く予想されるときのマルチサイクルカイネティクスの場合(キャプチャー法、リガンドが経時的に劣化する場合)
- Constant:Steady State Affinity Rmax constant のモデル式を使用する際に、ポジコンのRmax等から算出した検体のRmaxなどをconstantとして入力するときがあります。これは、高濃度帯で非特異的結合が起こる検体で平衡値(Affinity) 解析を用いた解析をしたい場合に適用されます。
③ tc(流速に依存しないマストランスポート定数)
- 通常globalにして解析します。こちらも添加濃度やサイクル毎の環境などの違いに理論的には依存しない値です。
④ RI(図2参照)
- localかconstant=0のどちらも利用されます。(Biacore™ T200, S200, X100ではlocal、Insightではconstant=0がデフォルトの設定)
- 良好なアッセイセットアップの下で取得した良好なセンサーグラムでは、通常RIに相当するアナライト添加前後に瞬間的に上下する“段差”は生じません。従ってその場合どちらで解析しても取得したいka, kdの値にほとんど影響を及ぼしません。
- 何らかの理由でこの“段差”の相対的な大きさが無視できない場合はlocalにします。
-
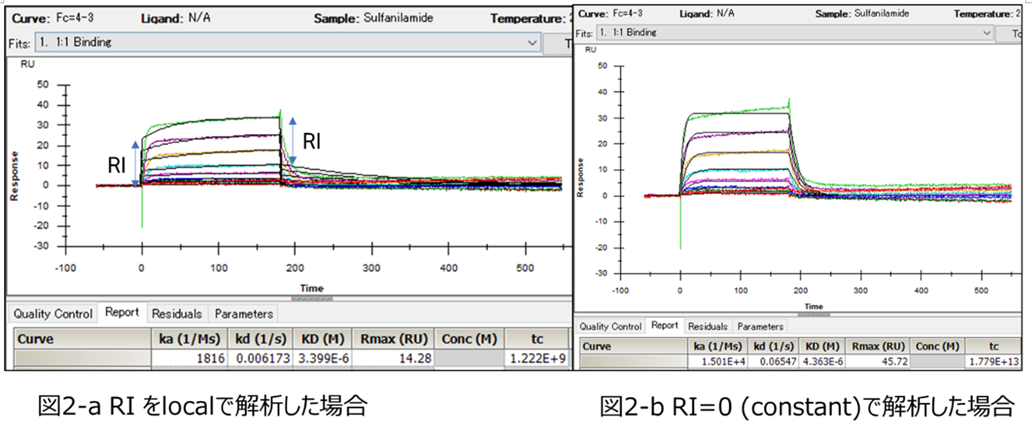
結合・解離が速いKinetics解析の場合、実際には見た目では“段差”が存在しない場合でも、計算上RIをlocalの変数にした方がChi2の小さいフィッティングになることがあります。その場合はどちらの設定にした方が、測定したいka, kdの真値を反映したフィッティングになるのかを目視で判断する必要があります。実際に低分子化合物Sulfanilamideの相互作用データを基にご説明したいと思います(図2)。
このデータは(おそらくベースラインの再現性が悪く)解析対象のセンサーグラムの解離相がベースラインまで落ち切らない形状をしていて完ぺきとはとは言えないアッセイセットアップで得られたセンサーグラムといえます。図2-aではRIをlocal、図2-bではRIをconstant=0でフィッティング解析しています。どちらもChi2の大きさとしては同程度のフィッティングでしたが、ka, kdは大きく異なっているのが分かると思います。
図2-aのフィッティングの方は速い結合解離形状部分をRIと認識して、測定したいSulfanilamideの相互作用部分は遅い結合解離のセンサーグラム成分の方としてフィッティングしています。ですが、目視で確認すると、本当のRIのような瞬間的な段差はないことに気づきます。
ですので、おそらくこの速い結合解離成分の方が、実際のSulfanilamideの相互作用を反映していて、遅い相互作用成分は理想的でないアッセイセットアップが引き起こした実験誤差である可能性の方が高いと思われます。
そこで図2-bの様にRIをconstant=0に設定してフィッティングすると図中のようなka, kdが算出されます。このka, kd は文献値*1であるka=2.3 ×104, kd=0.13と比較的近い値です。
*1D.G. Myszka / Analytical Biochemistry 329 (2004) 316–323
(補足)実際にこのデータをフィッティングする場合には不要に長く誤差の大きい解離部のセンサーグラムをカット(Remove selection)して解析するのも良い方法です。
⑤ drift(ベースラインドリフト、Insightのみ)
- constant=0がデフォルトの設定です。良好なアッセイセットアップの下で取得した良好なセンサーグラムでは、ベースラインドリフトはほとんどないはずという理由からです。従って、より信頼性の高いka, kdを求めたい場合はアッセイセットアップ上で(ダブルサブトラクション後の)ベースラインドリフトがない様にすることが優先されます。
- ドリフトを無くすことが難しい場合、あるいは無くさなくともka, kdの概算値が欲しい場合等にglobal またはlocalで変数として設定します。 相互作用プロファイルなどによって算出されるka, kdの誤差が大きくなることがありますので注意が必要です。
ここまではglobal、local、constantの使い分けをお話したのですが、もう少し発展させて、ここの使い方を工夫することで、Biacore™ 8K/8K+のような複数ニードルシステムの測定方法に広い選択肢を持たせることができる、というお話をしたいと思います。
これらのシステムはリガンド・アナライトをパラレルに流すことで単位時間当たりのデータ数を増やすことができますが、同時にチャネル間のばらつきが出てきます(装置設計上ばらつきは低くなるようにしているものの)。
そこで、Biacore™ 8K/8K+での便利な測定方法である Parallel Kinetics や 2D kinetics(図3)では、Rmaxをlocalにすることで解析を可能にしました。
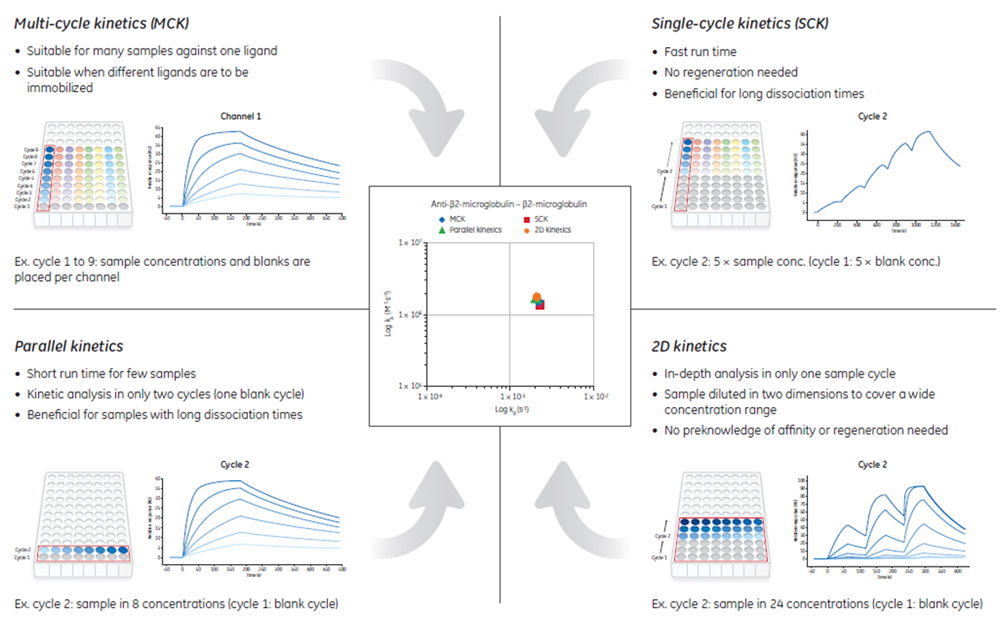
図3:
2.のinitial value (初期値)ですが、まずフィッティング計算の下記のイメージ動画(図4)をご参考ください。よくフィッティング計算はこのように転がるボールのイメージで説明されます。ボールはその空間の中で最も低いところに転がっていき、停止した場所がフィッティング計算の終了した“解”に当たる場所だ、という説明です。
この動画では空間の中で凹んでいるところは2か所存在し、最も深い場所にボールは停止しやすいですが、ボールを最初に置く場所(=初期値)によりもう一つの凹んでいる場所に停止することもあり得ます。
そのような場合、転がり始めの場所が適切でないと本来知りたい相互作用を反映した数値でないところで停止する場合もあります。Biacore™測定において、このような空間の全体像を測定者が認知することはできませんので、通常はデフォルトのinitial valueでフィッティングしたときに“あまりにも異常な解が算出された”時にInitial valueを変更して再解析する、ということになります。
異常な解と言えないときも、もしかしたら近接した場所に解(凹んでいる場所)が複数存在する可能性を完全に否定することはできませんが、経験上マストランスポート(MTL)が軽度で、アナライトの添加濃度範囲・時間も適切なセットアップをしているデータを取得していれば、Initial valueを想定の範囲で変更したところで同一の解が出ることがほとんどかと思います。
もしinitial value を変更して、近接値の解が得られることがあったとしたら、まずは上記のセットアップが適切か確認してみてください。適切と思われるのにやはりそのような現象がみられる(稀かと思いますが)としたら、複数の測定条件や繰り返し測定により真の解を明らかにしていくことになります。

図4:
今回はフィッティングのParameter settingの設定方法からその意味等をお話いたしました。
あまり目立たない小さな“表”ですが、突き詰めてみると意外と深い部分があるのをお伝えできたとしたら幸いです。