RUはResonance Unitの略で1000RU = 1ng/mm2という関係式がある。そのように理解している方はたくさんいらっしゃると思います。これはほとんど大きな間違いにはなりませんし、理解を簡単にするために良い関係式ではあるのですが、Biacore™コンシェルジュ愛読者の皆様にはもう少し深いお話をしたいと思います。
これにより、理論的RmaxやBinding level screenの誤差などについてわかるようになります。
1000RU=1ng/mm2の関係式についてもしかしたら少しびっくりする方もいらっしゃるかもしれませんが2つほど真実をお話したいと思います。
- そもそもRUの基になっているSPRの検出原理では密度変化を検出しているわけではありません。
- ka、kd、KDを算出するにはこの関係式は使われません。
まず1.についてお話します。Biacore™の検出原理となっている表面プラズモン共鳴(SPR)において、反射光が最も弱くなる入射光の角度(プラズモン共鳴角、1000RU=0.1°の変化)は、センサーチップ表面における金属の種類や入射光の波長やセンサ―チップ表面近傍の数100nm内の屈折率で決まります。Biacore™の場合、センサ―チップで用いられている金属は常に金ですし、入射光の波長も固定(一定の範囲の角度を“楔形”に入射しています)です。従いましてRU変化を決定づけているのは“屈折率変化”ということになります。(図1)そして、この屈折率変化は少なくとも溶液状態であれば密度(濃度)と比例関係にあることの想像がつくかと思います。ではBiacore™のセンサーチップ上ではどうなのでしょうか?そのあたりも含めて2.のお話に進みたいと思います。
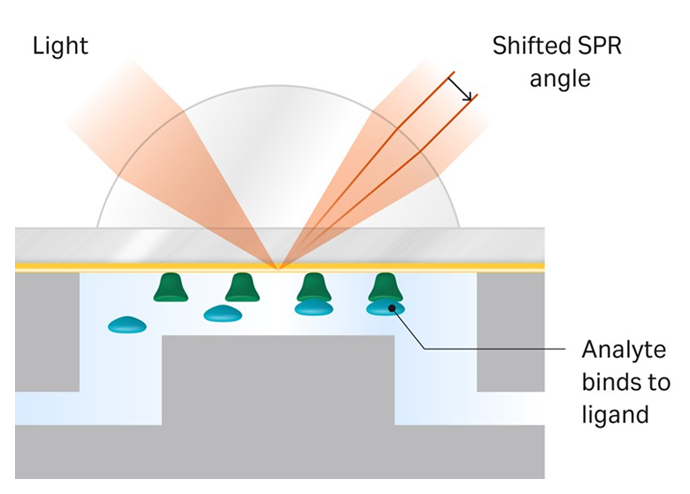
図1
次に2.の点ですが、これは例えばリガンドAとアナライトBとの相互作用を固定化量の異なる固定化量で測定する場合を考えるとイメージしやすいかもしれません。固定化量の異なるセンサーグラムはこの場合、レスポンスの高さは異なるのですが、相似形となり、また同じ分子A、Bを測定しているわけですので同一の解( ka、kd )を算出します。ここで、“レスポンスの高さが異なる”、というのが固定化量でなく、仮に1000RU=2ng/mm2と定義された装置で測定して起こったとしても、やはり1000RU=1ng/mm2と定義された装置で測定した場合と“相似形”になるはずですので、同じka、kd、KDになるわけです。
逆に言いますと1000RUが1ng/mm2だろうが、100ng/mm2だろうが、同じリガンドとアナライトの組み合わせであれば、同じka、kd、KDが算出されます。
これらの算出に重要なのは、RUがセンサーチップ表面上の密度に対して比例関係にあることであり、これは1990年の初代Biacore™を開発した当時のスウェーデンの研究チームが、研究開発時の重要なマイルストーンとなるデータとして取得しています1)。この時の手法はセンサーチップ上のRIラベルし各種タンパク質で密度とBiacore™でのRU変化の相関を取ることで行い、比例関係であることを確認し、そのいわゆる“副産物”として1000RU=1ng/mm2という相関式が実験的に生まれた、というわけです。(余談ですが、この報告のFirst AuthorのUlf Jonnsonは後のBiacore社のCEOになっています。)
では研究上この1000RU=1ng/mm2の関係式は全く使われないのか?といわれるとそんなことはありません。例えば代表的なところでは、以下の2点で使われます。
- A.理論的Rmaxを計算するとき
- B.レポートポイントを用いたスクリーニングをするとき(Binding Level Screen)
A.の理論的Rmaxの計算方法は2021年4月号の記事“Rmaxのトリセツ”でも触れた式で算出されます。この式は、リガンドとアナライトの単位密度あたりの屈折率が等しい(つまり1000RU=1ng/mm2で一定値)ことが前提となっています。実際タンパク質の同志の場合概ねそれで正しく、その前提を検証した論文なども書かれています2)。
先ほどのBiacore™の研究チームの報告の時代ではBiacore™で測定する目的はほとんどタンパク質だったので上記の記事内の式で良いのですが、現在の様に日常的に低分子を測定するとなるといくらかその前提となる“等しさ”の幅(つまり誤差)が大きくなります。先ほどのタンパク質での屈折率分布を検証した論文と同じように今度は実際の低分子化合物で示差屈折率計を用いて測定した論文3)なども報告されています。
それによると概ね2倍程度の単位密度あたりの屈折率の幅があるようです。従って低分子アナライトのRmaxを議論する場合はそのくらいの誤差がある可能性を気に留めておくのも良いかと思います。つまり(あまり実際問題として多く起こることではありませんが)、例えば理論的Rmaxが30RUの時に実測Rmaxが45RUだった場合、これを非特異的結合が乗っているとは言い切れない、ということです。
非特異的結合が起こる場合はセンサーグラムの形状も大きな判別のヒントになりえますが、もしきれいな特異的相互作用の形状だった場合、何らかの複合体構造やストイキオメトリーに関する情報と組み合わせて判別することになります。
B.のBinding level Screeningも低分子化合物の場合には一定の誤差に関する留意が必要と思われます。
Biacore™の現在のほとんどの解析ソフトウエアでは各化合物の結合レスポンス[RU]を分子量で割って、ノーマライズする機能がついています。これも各化合物の単位密度あたりの屈折率が等しいという前提条件の下計算されています。
従って先ほどの様に化合物の屈折率が異なる想定の元では一定の誤差が実際には含まれるということになります。つまり化合物の場合Binding level screenのランキング = アフィニティのランキングに必ずしもならないということです。
ただし、現実の測定のワークフローでは、スクリーニング後にはアフィニティ解析になりますので、その時に正確なランキングや数値が分かる、ということになります。またBinding level screeningの時には往々にして非特異的結合が含まれる化合物もいくらか存在することが多く、本質的に擬陽性は一定数出てくるのが普通です。
従ってBinding level screenでは、擬陽性・偽陰性はある程度出てくることを前提として、Threshold設定をするときに、スクリーニングからキャラクタリゼーションの戦略の中で、今回の屈折率の誤差も考慮に加えてみるのも一案です。
参考文献
- 1)U. Jonsson et. al., Advances in Biosensors 2, 291, 1992.
- 2)H. Zhao et. al., Biophysical Journal, 100, 2309, 2011.
- 3)T. M. Davis et. al., Analytical Biochemistry 284, 348, 2000.