AffinityとAvidity
この記事を読んでいただいている皆様はすでにご存じのことが多いかもしれませんが、まず最初に何故二量体分子はリガンド側にした方が良いかという理由をお話します。
Biacore™で求めるka、kd(KD)は1:1相互作用環境(Affinity環境)での結合・解離速度や結合の強さを決める定数になります(図1 左下式)。ところが、二量体をアナライトとすると、二量体の1本の“手”で結合している成分と2本の手が結合している成分が混在した環境(Avidity環境)になります。その環境でka、kd (図1右下図ではka1、kd1)を求めるにはaffinity環境で求めることに比べて誤差が大きくなるリスクが高くなります。これがなるべく二量体をリガンドにするのを多くの場合第一選択にする理由です。
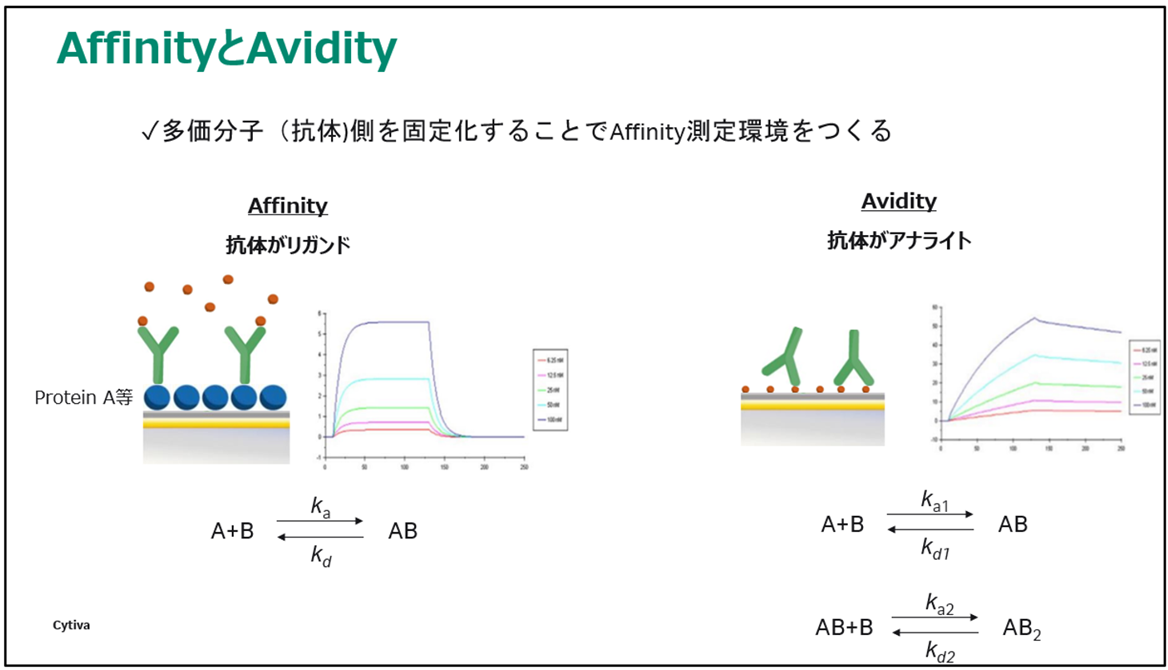
図 1:Affinity と Avidity
二量体アナライトでもAffinity環境?
さて、それではここで図1の右のセンサーチップ上のリガンドとアナライトの絵に注目してみたいと思います。上述の通り、この絵では1本の手で結合しているアナライト(抗体)と二本の手で結合しているアナライトが混在しています。
もし、リガンド間の距離が二本の手が届かないほど離れていればどうなるでしょうか。結合しているアナライトは1本の手だけですべて結合するはずです。
つまりリガンドの固定化量を下げるとどこかで1:1のaffinity の環境を作ることができる可能性があります。それを実際に行ったのが図2.の結果です。上図 6つのセンサーグラムはすべて同一のサンプル(リガンド:抗原、アナライト:IgG)ですが、固定化量(Rmax)が左から右に行くにしたがって小さくなっています。
それに従ってセンサーグラムの形状が変化していますが、全てに1:1 binding model式をフィッティングしてKD値を算出してRmaxごとにプロットしたのが下図になります。おおむねRmax=4RU以下からKD値が収束して、1:1のAffinity環境になることが示唆されます。

図 2:二量体(IgG)をアナライトとした測定で抗原固定化量を下げていった場合の変化
実際の測定手順について
前節のRmax=4RUとなるような固定化量という条件は、一般化できるほどのまとまったデータはありませんし分子量によっても異なってくるはずですので、それぞれのサンプルにおいて固定化(キャプチャー)量条件を多点でとって、検証する必要が出てきますので、相応のCycle数が必要になる測定になるはずです。
一方で、近年のBiacore™ 装置は上述のデータの装置より高感度(低ノイズ)な機種も市販され、測定成功の可能性をあげられるかもしれません。また、こちらの記事「このセンサーチップご存じですか?SensorChip PEGとSeries S SensorChip NA」でも簡単に触れましたが、2D表面構造を持つセンサーチップPEGを用いて、よりきれいなセンサーグラムを取得してきわめて低レスポンスにも関わらず信頼性の高い算出値を得たような例もあります。
簡単にできる測定とは言えないかもしれませんが、二量体分子をアナライトにしたい場合には一つの選択肢と言えます。