Dr. 近藤のコラム 2D-DIGEの熱い心
「二次元電気泳動は何をみているのか?」(1/3ページ)
二次元電気泳動の謎
二次元電気泳動法で観察されるタンパク質スポットがどのようなタンパク質に対応しているのかは、タンパク質スポットを回収してタンパク質同定の実験をしてみなくては分からない。タンパク質同定の実験が難しかった時代はどんなタンパク質がみえているのかあまり分からなかったので二次元電気泳動法に過剰な期待が寄せられていたようである。やがてタンパク質同定の実験がある程度普及してくると、期待していたようなタンパク質が見つからなかったことから多くの研究者は二次元電気泳動法に見切りをつけるようになった。そしていわゆる「post genome era -プロテオミクスの時代-」になるとタンパク質同定実験は以前とは比べものにならないほど簡単になった。しかし二次元電気泳動法ではどのようなタンパク質がみえているのか、誰も知らない。今もって謎である。
通常の比較実験ではサンプル同士を比較し濃度差のあるタンパク質スポットだけがタンパク質同定実験にまわることになる。そうすると、何かの刺激や状況の変化に反応して濃度が変化しやすいタンパク質スポット、少々実験のぶれがあっても濃度差が再現性よく得られるタンパク質スポット、ゲルの中央にあったり他のスポットからよく分離されるタンパク質スポット、発現量や分子量やアミノ酸の内容から同定実験が成功しやすいタンパク質スポット、などが発現実験の結果として報告される。その結果、同じようなタンパク質が何度も何度も繰り返し学会や論文で報告されることになる。その代表格がヒートショックタンパク質、細胞骨格タンパク質、ある種の代謝系の酵素、などである。このようなことから、あたかもこれらのタンパク質しか二次元電気泳動法では観察されないかのような錯覚をいだいている研究者は多いのではないだろうか?
二次元電気泳動法の限界を探る試み
二次元電気泳動法でどのようなタンパク質が観察されるのかをテーマとした論文としては次のようなものがある。
- Gygi SP, Sorthals GL, Zhang Y, Rochon Y, and Aebersold R, Evaluation of two-dimensional gel electrophoresis-based proteome analysis technology. Proc. Natl Acad Sci USA, 2000, 97, 9390-5.
この論文ではコドンバイアスの低い遺伝子を発現量の低い遺伝子と仮定し、二次元電気泳動法のある特定のゲル範囲について50個のスポットを同定実験に選択した。二次元電気泳動法ではコドンバイアスが低く発現量が低いと予測される遺伝子産物は観察されないこと、コピー数の少ないタンパク質を観察するには、50 mgという大量のタンパク質を添加しないといけないこと、などを結論づけている。ちなみにファーストオーサー、ラストオーサーは質量分析を使ったプロテオーム解析の第一人者である。
この論文はゲノム情報も使用したなかなか本格的な実験である。ゲルのパターンはきれいだし、統計的考察も行われている。二次元電気泳動法の限界についてはこれで決着がついたかのように思われた方もおられるかもしれない。しかし、よく考えてみると実はそうではない。
二次元電気泳動法で観察されるタンパク質の種類は、使用するサンプルの種類、タンパク質の抽出法、電気泳動の方法、タンパク質の検出法、などによってがらりと変わってしまう。前述のGygiらの論文では、イーストを出発材料としてタンパク質を抽出し、自作の4.5-5.5の狭小幅のイモビラインゲルで一次元目、10%のホモジニアスゲルで二次元目の分離を行ったうえで銀染色を使ってタンパク質スポットを検出している。しかしこの方法が二次元電気泳動法のすべてではないことは言うまでもない。組織・臓器によって含まれるタンパク質の種類や発現量のバランスは異なるし、サンプル調製の際に分画をとることによってゲル画像はまったく違うものになる。一次元目の電気泳動法もIPGを使うものもあればアガロースを使うものもあり、等電点の幅やゲルの長さもさまざまであり、それぞれに異なるタンパク質を観察することになる。二次元目の電気泳動もアクリルアミドの濃度やゲルのサイズを変えることによって観察されるタンパク質は異なってくる。検出法も、クマシー染色、銀染色、蛍光色素染色などに加え、生きた細胞を35Sや32Pでアイイソトープ標識する方法、2D-DIGEのようにリジンやシステインを標識する方法、リン酸化タンパク質や糖鎖修飾を受けたタンパク質だけを蛍光色素で染色する方法、さらには抗体を使用する方法など、じつにバラエティーに富んでおり、それぞれに異なるタンパク質が観察される。これらのステップの組み合わせは、一人の研究者の一生の実験量を考えれば、現実的には無限と言ってもいいくらいの数になる。したがって、ある実験プロトコールで観察されたタンパク質の種類がどうであっても、そのデータをもとに二次元電気泳動法で「一般的に」どのようなタンパク質が「観察されない」のかを論じるのは妥当とは言えない。
それではどのような方法で二次元電気泳動法の性能を評価するのがいいのだろうか?二次元電気泳動法で観察できる可能性のあるタンパク質あるいは観察できる可能性が低いタンパク質を議論するためには、理屈のうえではあらゆるプロトコールを使った実験を行って網羅的にタンパク質を同定する必要があることになる。しかしそのような実験自体は医学・生物学研究的には何の発見にもつながらないだろうから、何か目的を見いだせない限りなかなかやる気になれない。
タンパク質データベースからみる二次元電気泳動の可能性
本稿ではまったく別の視点から二次元電気泳動能法の可能性と限界を考えてみる。実験をすることなく「手軽に」かつ「ゲノムワイドに」ということころがポイントである。利用するのは公開されているヒトタンパク質のデータベースである。公開されている情報をもとにして、ゲノムワイドにはタンパク質はどのような分子集団なのか、二次元電気泳動法ではプロテオームのどの部分が観察できる可能性があるのかを考察する。
翻訳後修飾などでタンパク質の分子量や等電点は変化するのでアミノ酸配列からの予測はかならずしも現実とは一致しないのだが、ゲノムワイドに二次元電気泳動能の可能性を検討するうえではタンパク質の配列データベースの情報を用いるのは得策である。使用したデータベースはUniProtKB/Swiss-ProtとUCSC GoldenPath proteinDBである。これらのデータベースに収録されているタンパク質をプロテオームの全体像と仮定し、二次元電気泳動法はプロテオーム全体像のどこをみることができるのかを考える。データ分析は三井情報株式会社の宮田彩香氏そしてCytiva社の平野穣氏にご尽力いただいた。
アミノ酸残基の視点からみる二次元電気泳動法の可能性
まずは2D-DIGE法について。2D-DIGE法で使われる蛍光試薬は2種類、CyDye™ DIGE Fluor minimal dyeとCyDye™ DIGE Fluor saturation dyeである。それぞれ、リジン残基とシステイン残基をラベルして観察する。つまり、前者を用いた場合はリジン残基を、後者を用いた場合はシステイン残基をもたないタンパク質は観察できない。網羅的にタンパク質を観察するプロテオーム解析で、解析できないタンパク質が存在するとしたら問題である。しかしこの問題はどれくらい重大なのだろうか?すなわち、どれくらいのタンパク質がDIGE技術で観察できないのだろうか?
UniProtKB/Swiss-Prot Protein Knowledgebase Release 56.2 Statisticsによると、収録されているタンパク質の中でリジン残基は8番目に出現頻度が高く、システイン残基は2番目に出現頻度が低いアミノ酸残基である。ああそう、という感じで、このデータからは特に何かが分かるわけではない。
リジン残基、システイン残基を持たないタンパク質を検索するためにUniProtKB/Swiss-Prot (Release 14.2 - Sep 23, 2008)に収録されているヒトタンパク質20,328種類を調べた。配列を含むファイルをダウンロードし、マイクロソフトアクセスを使って検索をかけたところ、リジン残基を含まないタンパク質は118個、システイン残基を含まないタンパク質は579個存在することが分かった。それぞれ、全体の0.58% (118/20,328個)および2.85% (579/20,328個)に相当する。
すなわち、リジン残基やシステイン残基をもたないタンパク質は確かに存在するものの、全体に占める割合はきわめて少ない。両者合わせてもわずか3.43% (0.58+2.85=3.43%、118+579=697個)に過ぎない。DIGEの蛍光色素の特異性のために観察できないタンパク質が存在するというのは、それほど大きな問題とは言えないのではないかというのが筆者の感想である。
等電点の視点からみる二次元電気泳動法の可能性
一次元目の電気泳動は等電点にしたがってタンパク質を分離するので、タンパク質は等電点に関してどのような分布状況にあるのかは興味深いところである。あまりタンパク質が存在しない等電点領域よりは、「タンパク質密集地帯」を調べる方が発現差のあるタンパク質を発見できる確率が高いだろう。下の図は18,729個のタンパク質を対象として等電点を横軸にとり、縦軸にそれぞれの等電点をもつタンパク質の数をプロットしたものである。出典はUCSC GoldenPath proteinDB(proteins070202)である。一目でわかることは、等電点7あたりに頻度的に落ち込みがあることである。この付近の等電点をもつタンパク質は、生体内のpHがだいたいこの辺りなのでたまたま沈殿しやすく、進化の過程で淘汰されたのかもしれない。
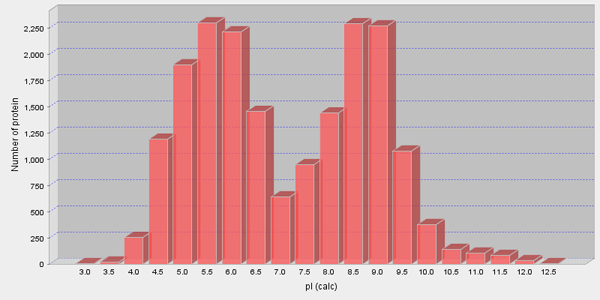
二次元電気泳動法の実験を考えるうえでこのデータから言えることは、等電点4から10付近を調べることが効率的であるということである。逆に、等電点が4より小さいタンパク質(23個、0.12%)や等電点が10より大きいタンパク質(719個、3.8%)を調べるには何か特別な目的が必要だろう。
市販されているIPGゲルは等電点3から10をカバーしている。狭小幅のIPGゲルもこの範囲で市販されている。ちなみに筆者のラボでは等電点4から7のIPGゲルを専ら使用している。等電点幅が4から7のIPGゲル、そして等電点幅が6から10の市販IPGゲルを組み合わせて等電点幅4から10を調べれば、17,987個(96.0%)のタンパク質が観察できることになる。普通に網羅的に調べたいのであればこれで十分ではないだろうか。
→続き(分子量の視点からみる二次元電気泳動法の可能性)を読む
 DIGE道場 第6回にもどる
DIGE道場 第6回にもどる
 DIGE道場 トップページ
DIGE道場 トップページ
近藤 格 先生の研究室 Webサイト
2D-DIGE に関する基礎情報はこちらから

