Dr. 近藤のコラム 2D-DIGEの熱い心
「二次元電気泳動は何をみているのか?」(2/3ページ)
←前のページに戻る
分子量の視点からみる二次元電気泳動法の可能性
一次元目の電気泳動法にイモビライン、二次元目の電気泳動にSDS-PAGEを使用する場合、だいたい10から200 kDaの範囲のタンパク質が観察される。この10から200 kDaという範囲にどれくらいのタンパク質が含まれているか、ということについてタンパク質のデータベースを調べてみた。
まず、公開されている統計データとしてはUniProtKB/Swiss-Prot Protein Knowledgebase Release 56.2 statisticsがある。ここにはタンパク質の大きさと頻度の関係がグラフで記載されている。このページでは横軸にタンパク質の長さ(アミノ酸の個数)、縦軸に度数をとったグラフが「Length distribution of the sequences」として掲載されている。これによると、アミノ酸にして800-1000個くらい以下の長さのタンパク質が多いこと、極端に長いタンパク質は少ないことが分かる。しかしアミノ酸の数で言われても分子量に換算しないとピンとこない。
アミノ酸の数ではなく分子量についてプロットしたのが下記の図である。これはUCSC GoldenPath(proteins070202)に収録されているデータファイルをもとに計算したものである。
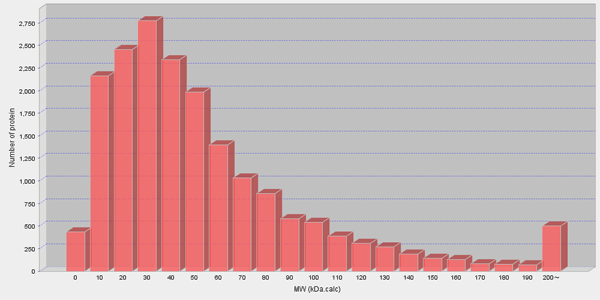
データベースに登録されている 18,729個のタンパク質のうち、分子量が10から200 kDaのタンパク質は17,794個存在し、これは全体の95.0%に相当した。逆に、分子量が10 kDaより小さいタンパク質は436個、分子量が200 kDaより大きいタンパク質は499個、存在した。パーセンテージにすると、それぞれ2.3%および2.7%に相当する。
通常のSDS-PAGEではなくバッファー系にTris-Tricineバッファーを使うことで10 kDa以下のタンパク質を観察することが可能である(文献1)。北里大学の大石先生たちが行っているように、一次元目の電気泳動にアガロースゲルを使うことで200 kDaを超えるタンパク質をカバーすることも可能である(文献2)。しかし、取り敢えず全体を観察したいという研究者であれば、IPGゲルと通常のSDS-PAGEをまずは試してみるのが効率的のようだ。
文献
- Schägger H, von Jagow G. Tricine-sodium dodecyl sulfate-polyacrylamide gel electrophoresis for the separation of proteins in the range from 1 to 100 kDa. Anal Biochem., 1987 166, 368-79.
- Oh-Ishi M, Maeda T. Disease proteomics of high-molecular-mass proteins by two-dimensional gel electrophoresis with agarose gels in the first dimension (Agarose 2-DE). J Chromatogr B Analyt Technol Biomed Life Sci., 2007 849, 211-22.
仮想二次元電気泳動法からみる二次元電気泳動法の可能性
計算上の等電点と分子量から仮想二次元電気泳動ゲル画像を作成すると下記のようなものになる。前述の等電点と分子量のデータを合わせたものである。赤枠で囲ってある領域は筆者がルーチンに2D-DIGE法で観察している領域である(等電点4~7のIPGゲルとSDS-PAGE)。
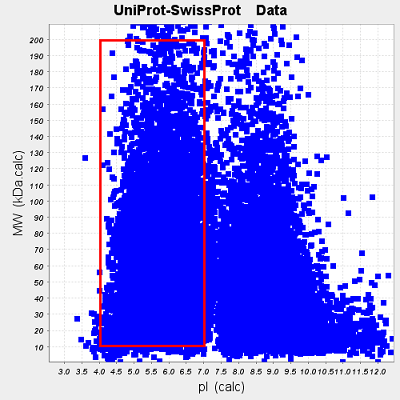
等電点幅4~7のIPGゲルと通常のSDS-PAGEゲルの組み合わせで観察できる可能性のあるタンパク質は8,810個、これは全体の47%に相当する。また、等電点幅6~10のIPGゲルを使った場合だと、観察できる可能性のあるタンパク質は11,841個、全体の63.2%となる。両者を組み合わせることで、17,133個(91.5%)のタンパク質が観察できる可能性がある。
仮想二次元電気泳動画像は以前の論文でも報告されている(文献1, 2)。等電点7付近の落ち込みは同様に報告されている。等電点7付近の頻度の落ち込みは生物種を問わず一般的に言えることなのかもしれない。
文献
- Medjahed D, Smythers GW, Powell DA, Stephens RM, Lemkin PF, Munroe DJ, VIRTUA2D: A web-accessible predictive database for proteomics analysis. Proteomics, 2003, 3, 129-38.
- VanBogelen AR, Abshire ZA, Moldover B, Olson RE, and Neidhardt CF. Escherichia coli proteome analysis using the gene-protein database. Electrophoresis, 1997, 18, 1243-51.
翻訳後修飾の視点からみる二次元電気泳動法の可能性
同一のタンパク質が複数のタンパク質スポットとして観察されるのが二次元電気泳動法の特徴である。この現象は翻訳後修飾を反映していると一般には考えられている。筆者が構築中のプロテオームデータベースであるGeMDBJ Proteomicsに登録されているタンパク質スポットのうち、LC-MSMSとマスコット検索で得られた同定情報が付加されているものは今のところ1,095個である。マスコット検索1位として同定されたタンパク質の種類としては443種類に対応する。同じタンパク質が複数のタンパク質スポットで同定されたので、タンパク質の種類数はタンパク質スポットの数の約40%である。同定するタンパク質スポットの選択はかなりランダムに行っているので、タンパク質の前処理をしない場合に限ればこの繰り返しの傾向はかなり一般化できると考えている。
この繰り返しの頻度が一般にあてはまるとしよう。アイソトープを使って定量性と網羅性をアピールしている最近の質量分析の論文では4,399種類のタンパク質が同定されている(文献1)。数だけかせぐのであればもっとたくさんの種類のタンパク質が観察されるのだろうが、アイソトープを使って定量的にとなるとこのくらいの数が限界のようである。このタンパク質の数に匹敵するタンパク質スポットを繰り返し頻度から逆計算すると10,873個、すなわち約1万個となる。「1万個」という数は、前分画なしに二次元電気泳動法で観察されるタンパク質スポットの上限のように語られることが多い(一つの例として文献2)。すなわち2D-DIGE法を網羅的に行えば最新の質量分析実験と変わらない網羅的なデータがとれることになる。
1万個を超える数のタンパク質スポットを観察するにはどうしたらいいだろうか?一つの方法はゲル面積を増やすことである。なぜか?一つのタンパク質スポットには複数のタンパク質が含まれていることが従来から報告されている(文献3, 4)。筆者も経験するところであるが、複数のタンパク質が一つのスポットから同定されることから、同定実験の結果は一義的に解釈できないことがある。この問題はゲルの分離度を上げることで解決できる。複数の遺伝子産物が小数点何桁かまでたまたまぴったり同じ等電点と分子量をもつことは考えにくいので、分離度を上げていけば今まで一つのスポットに見えたものが複数のスポットに分離される。ゲル面積を増やすことでスポット数が増加することは以前から報告されていたが、その背景を説明しているのが同定実験における質量分析のこういったデータである。泳動距離が2倍のゲルを使用するとスポットの数も約2倍に増加することを筆者も経験している。これは画像解析ソフトの認識能力にも関係する。解像度が高いゲルだと画像解析ソフトが再現性よくスポットを認識するので、結果的に比較解析の実験に使用できるスポットの数が増加する。試算では、2D-DIGE法でスキャナーの面積が許す限り大きなゲルを使ったり、複数のIPGゲルを組み合わせたりすると、観察できるタンパク質スポットの数は1万個を余裕で超える。
文献
- de Godoy LM, Olsen JV, Cox J, Nielsen ML, Hubner NC, Frohlich F, Walther TC, Mann M. Comprehensive mass-spectrometry-based proteome quantification of haploid versus diploid yeast. Nature, 2008 Sep 28.
- Gygi SP, Sorthals GL, Zhang Y, Rochon Y, and Aebersold R, Evaluation of two-dimensional gel electrophoresis-based proteome analysis technology. Proc. Natl Acad Sci USA, 2000, 25, 9390-5.
- Campostrini N, Areces LB, Rappsilber J, Pietrogrande MC, Dondi F, Pastorino F, Ponzoni M, Righetti PG. Spot overlapping in two-dimensional maps: a serious problem ignored for much too long. Proteomics, 2005, 5, 2385-95.
- Gygi SP, Sorthals GL, Zhang Y, Rochon Y, and Aebersold R, Evaluation of two-dimensional gel electrophoresis-based proteome analysis technology. Proc. Natl Acad Sci USA, 2000, 25, 9390-5.
膜タンパク質、糖鎖修飾タンパク質、難溶性タンパク質は二次元電気泳動法で観察できるのか
「膜タンパク質は二次元電気泳動法で観察できますか?」という質問を講演後にいただくことがある。膜タンパク質は二次元電気泳動法では観察されにくいと言われている。糖鎖修飾されたタンパク質はIPGゲルに入りにくいという説もある。溶かすことができないタンパク質や電気泳動中に沈殿してしまうタンパク質も二次元電気泳動法には向かないと聞く。本当だろうか?
これらの質問に対する答えを筆者はもっていない。確実に発現していると分かっている膜タンパク質や糖鎖修飾タンパク質を適当にランダムに数100個ほど選んで二次元電気泳動/ウェスタンブロッティングをしてみれば推測ができるかもしれない。難溶性タンパク質については、溶けないタンパク質は二次元電気泳動法では難しいような気がするが、網羅性を犠牲にしてよいのであれば方法があるかもしれない。
超微量タンパク質は二次元電気泳動法でどこまで観察できるのか
発現量がもっとも高いタンパク質と最も低いタンパク質を一枚のゲルで観察することは、検出系のダイナミックレンジやゲルの面積からして不可能だろう。例えばアクチンとp53が一枚のゲルで同時に観察できたりはしない。何かしらのタンパク質の前分画を行ったり複数のゲルを組み合わせたりする工夫が必要である。この際に問題なのが、「超微量」という特徴は「リン酸化タンパク質」「糖タンパク質」「膜タンパク質」などと違ってタンパク質の何らかの物理的性格を反映していないことである。すなわち量の少ないタンパク質、という選択基準でタンパク質を物理的に集めることができないということである。
発現量の少ないタンパク質を観察したいという場合、ただ量が少なければいいというのではなく、解析したいタンパク質群が漠然とあって、それらの発現量がたまたま低い、というケースが多いのではないかと推測している。具体的には、受容体やそのリガンド、転写因子、シグナル制御因子などである。機能的にグループを形成していたり、局在が共通だったり、配列に相同性があるようなタンパク質群である。このようなタンパク質は機能的に重要であると同時に発現量が低い。
ここで役立つのが特異的プローブである。特定のタンパク質ファミリーということであればその一群をある程度の特異性でプローブを使ってまとめて回収し、二次元電気泳動法に展開することは十分可能である。この周辺の実験法はいくらでもあって、二次元電気泳動法の多様性の元となっている。対象とするタンパク質はどのような共通性が、たとえばアミノ酸配列のレベルで存在していて、どのようにすればまとめて回収することができるか、というところから新しい実験のアイデアが発生する。たとえば、ある重要な共通ドメインをもつタンパク質をまとめて回収して二次元電気泳動にかける、という実験は今なら可能だろう。タンパク質の前分画は多くのバリエーションがあり、研究者が技術的な独自性を発揮するよい局面となっている。前分画のバリエーションに加え、他の技術にはない二次元電気泳動法ならではの長所、生物学的に意味のある分画を考えて実験を組み立てることがDIGEである。
ここまでのまとめ
- リジン残基、システイン残基をもたないタンパク質はそれぞれ全体の0.58%、2.85%しか存在しない。したがって、DIGE技術でほとんどのタンパク質が理論的には観察できる。
- 一次元目に市販IPGゲル(等電点4から7と等電点6から10)、二次元目にSDS-PAGEという一般的な二次元電気泳動法の実験条件では、分子量、等電点という視点からは全体の91.5%が観察されうる。
- 網羅性拡張にはゲル面積の拡大、タンパク質の前分画、複数のゲルの組み合わせがポイント。
→続き(別の視点からデータベースのデータを眺めると)を読む
 DIGE道場 第6回にもどる
DIGE道場 第6回にもどる
 DIGE道場 トップページ
DIGE道場 トップページ
近藤 格 先生の研究室 Webサイト
2D-DIGE に関する基礎情報はこちらから

