Sensor Chip SAの構造
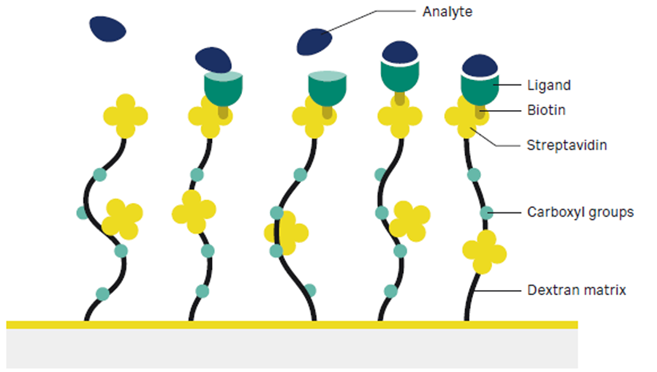
Figure 1 Sensor Chip SAの構造
Sensor Chip SAは、Sensor Chip CM3にあらかじめstreptavidin(以下、SAと呼びます)を共有結合させたものです。すなわち、デキストランがありますのでチップ表面は親水性ですし、カルボキシル基が残っておりますので中性緩衝液中で電離して負に荷電しています。SAの固定化密度や固定化方法は公開されておりませんが、納品時には非共有結合でチップ表面に留まるSAが残存しているため、利用前にチップ表面を洗浄するステップがあることには注意しなければなりません(後述します)。
リガンドはビオチン標識さえされていればマイルドな緩衝液でチップ表面に添加されるだけで非常に強烈な親和力(KDは約10-15 M)で結合します。非共有結合ではありますが、理論的には数か月に渡っての測定期間中に解離する量を考えなくとも良いくらいの親和力となるため、実質的には共有結合として取り扱って良いです。またこの強烈な親和力のためにSAからビオチンのみを解離させる手段はなく、したがって別のリガンドを付け直すといった操作はできません。
運用上の注意点
固定化方法と固定化量の見積り
固定化方法はそれぞれの装置にプリセットされたテンプレートをご利用いただくことをお勧めいたします。ここでは何を行っているか確認してみましょう。
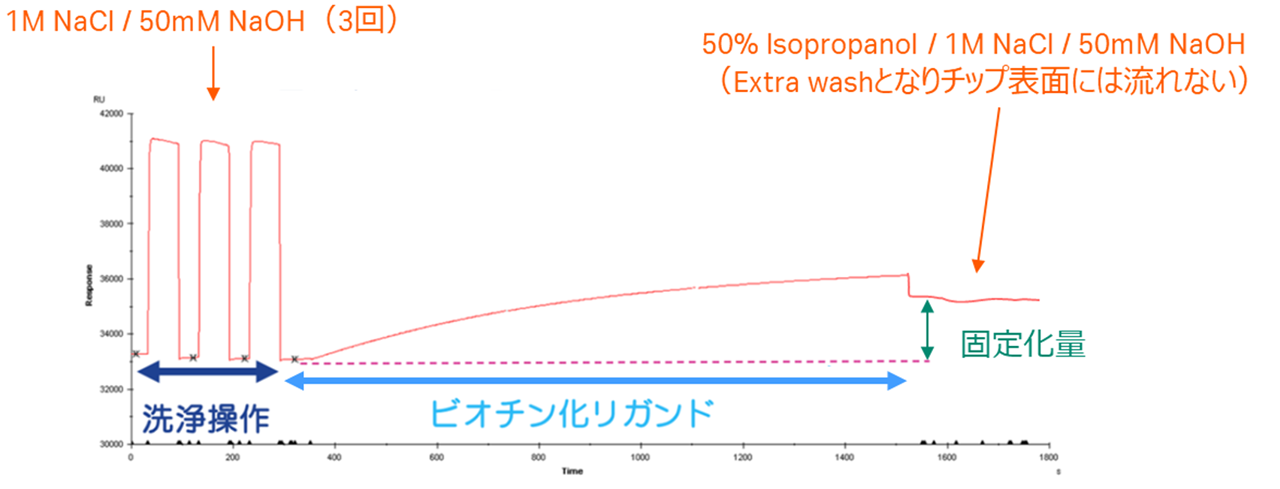
Figure 2 プリセットされたテンプレートを使用した際のSensor Chip SAの固定化のセンサーグラム(リガンドが核酸の場合)
流れとしては3ステップです。
- (洗浄操作)上述の通り納品時にはSensor Chip SAは非共有結合でチップ表面に留まるSAが残存しているため、3回の1M NaCl / 50mM NaOHの混合溶液でチップ表面を洗浄しています。
- (固定化)ビオチン標識リガンドが添加されます。
- (Extra wash)最後にセンサーグラムには現れませんが流路のみを洗浄する操作として50% Isopropanol / 1M NaCl / 50mM NaOHの混合溶液が添加されます。
ステップ1:洗浄操作
Figure 2をよくご覧いただくと、3回の1M NaCl / 50mM NaOHの混合溶液の添加によりベースラインが低くなっていることが分かります。これはまさしくSAが除去された様子です。測定中にベースラインが変動することを防ぐため、この操作はReference cellにも実施しなければなりません。
Biacore™ 1 seriesやBiacore 8™ seriesで用いられるInsight Control Softwareでは、Figure 3のようにReference cellにだけリガンドが添加されないMethodとなっていることが分かりやすく表示されています(Ligandの背景の青枠がFlow cell 1に入っていないことがご確認いただけると思います)。
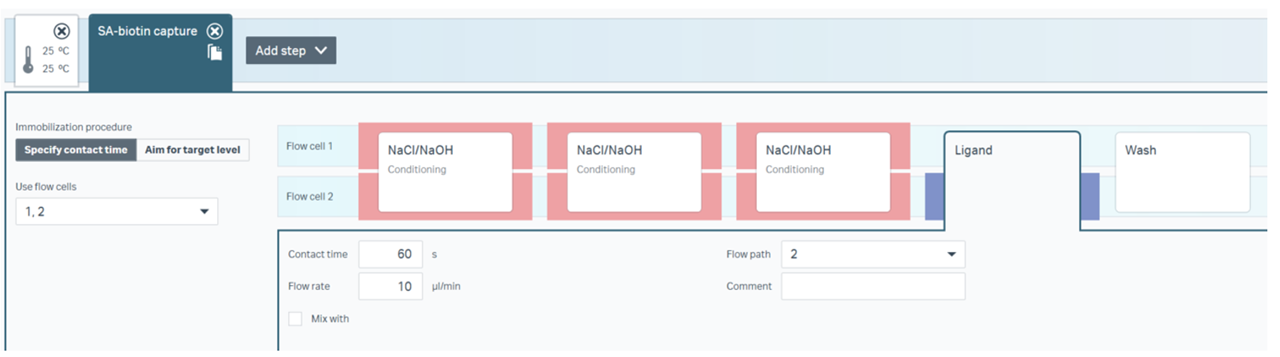
Figure 3 Biacore™ 8 seriesのプリセットテンプレート
続いてBiacore™ T200/S200/X100のテンプレートを見てみます(Figure 4)。
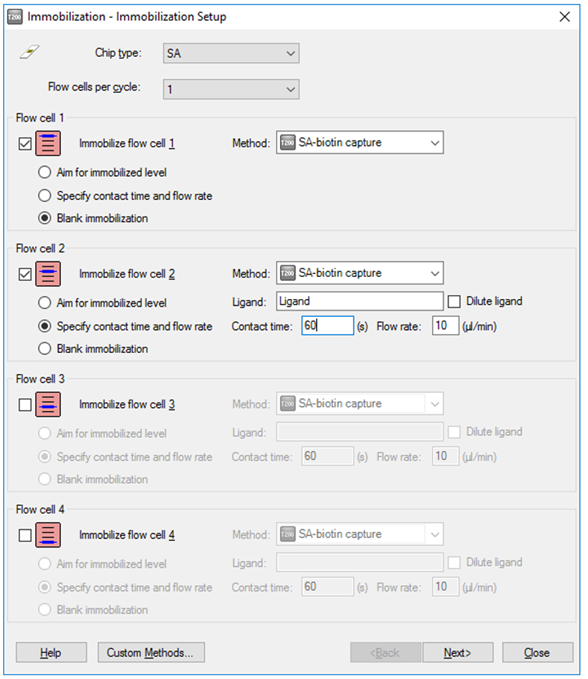
Figure 4 Biacore™ T200のプリセットテンプレート
この場合、Reference cell(Flow cell 1)に対してはBlank immobilizationを実行することで同様の処理を行うことが可能です。なお、下方のCustom Methodsをクリックすると、このプリセットされたテンプレートの詳細を確認することが可能です。
ステップ2:固定化
一般的な水系の緩衝液(HBS-EP+を推奨)において、ビオチン標識されたリガンドを添加するだけで固定化が進行します。サンプルの固定化量はResponse boundを採用します(改めてFigure 2をご確認ください)。サンプルのビオチン標識方法についてはこちらをご参照ください。経時的に活性化エステルが加水分解するアミンカップリング法とは異なり、SAチップに対する固定化は時間制限がありません。そのため初めて取り扱うサンプルの場合はとりあえず薄い濃度で固定化を実行し、足りなければ後で追加するという戦略を取ることができます(付け過ぎてしまった場合は外すことはできませんのでご注意ください)。どれくらい固定化すれば良いか、という議論はこちらでご確認いただくとして、そもそもどれくらいの固定化量が限界になるのかはある程度把握しておく必要があります。
リガンドがタンパク質の場合
タンパク質に対して後からビオチンタグを付与する場合、フリーのビオチンが除去されていることを必ずご確認ください(詳細はこちら)。その上で、数pM-数nM程度の濃度にて1分ほど添加すれば多くのケースですぐにサチュレートするはずです。CM5チップにアミンカップリング法で固定化する時とは異なり、最大固定化量は非常に小さくなります。例えば、一般的な抗体でしたらCM5チップなら8000-15000 RU程度の固定化量が稼げていたところ、SAチップですと数千RUが限度で、悪いと数百RU程度になるはずです。1分子に1ビオチン分子が導入されていることも重要です。1分子に多くのビオチン分子が導入されていると固定化量の減少につながります。
リガンドが核酸の場合
数nM-数十μM程度の濃度にて最長30分程度を目安に固定化を試みてください。しかし核酸自身が負電荷を帯びるため鎖長によってはチップ表面の負電荷と反発し、うまく固定化できない場合があります。あらかじめ静電的な反発を抑えるためリガンド溶液は500-1000mM NaClとして固定化を試みてください。最大固定化量は鎖長によりかなり変動しますが、20mer程度なら2500RU程度、50mer以上になると3000RU程度が目安となるでしょう。
高塩濃度条件ならば鎖長は最大で200nt程度まで固定化可能ですが、それでもうまくいかない場合は陽イオン性の界面活性剤CTABを利用した方法が有効な可能性があります。CTABを使用する場合の条件は、核酸濃度は10μM程度、溶媒は10mM Hepes(HBS-EP+バッファーではありませんのでご注意ください), pH7.4, 0.6-1mM CTAB程度で試します。CTABを使用する場合は、CTABの正電荷ミセルに核酸が取り込まれることで電荷による「濃縮効果」を得られることを期待しています。そのため、逆に低塩濃度条件の方が望ましくなりますのでご注意ください。
参考までに、負電荷が問題になる末端アミノ標識核酸をCTAB含有溶媒でアミンカップリングしている実験例は次を参照してください。
【Biacore】末端をアミノ化した核酸の固定化方法を教えてください。
ただし、こちらで示されているPDFの、ResultsのSensorgram1の文中の表記に誤りがありますのでご注意ください。
誤:0.6μM CTAB
正:0.6mM CTAB
Reference cellはビオチンでブロッキングしておくべきか?またActive cell上の未反応のSAはビオチンでブロッキングをしておくべきか?
結論から申し上げますとほとんどのケースでどちらも不要です。ブロッキングはベアなSAに対してアナライト分子が非特異的に結合してしまうことを避けることを目的にしますが、そうしたケース自体やや稀ですし、そもそもブロッキングの操作で完全にSAを覆うことは難しいためです。そればかりか、隣接するActive cellにキャリーオーバーし、リガンドの固定化量を低下させるリスクすらあります。SA自体と非特異的な結合をする場合はSensor Chip NAという代替品もご検討ください。
仮に実施の場合は50μM amino-PEO-biotinを使用します。ブロッキング後にも必ず流路の洗浄を実施してください。
ステップ3:Extra wash
リガンドを添加し終えると、センサーグラムには現れませんが流路のみを洗浄する操作として50% Isopropanol / 1M NaCl / 50mM NaOHの混合溶液が添加されます。ビオチン標識されたサンプルはどうしても疎水性が高くなりますのでシリコンゴムでできた流路に残留しやすい課題があります。これを除去するためにこうした有機溶媒の混合溶液を添加しています。Extra washは金膜の上は通らない操作になりますから、せっかく固定化したリガンドを破壊することはありません。
Biacore™ T200/S200/X100などではこのExtra washのコマンドはManual Runからは実行できません。したがって固定化時はWizardから実行することをお勧めいたします。なお、InsightではInteractive RunにてExtra washにあたるWashコマンドが実行可能ですので、一連の操作はManual Runで実行しても問題ありません。
思ったよりも固定化量が少なかった際に、追加で固定化したい場合
既にサチュレートしたセンサーグラムが得られている場合はあまり固定化量を増やすことはできないのですが、それでも試したい場合やリガンドの濃度が低すぎて固定化が進行しなかった場合には固定化を再度実施することが可能です。ただし、全く同じMethodを用いると、チップ表面に再び3回の1M NaCl / 50mM NaOHの混合溶液が添加されてしまうことになり、せっかく固定化したリガンドが破壊されてしまいます。解決方法としては単純で、この3回の1M NaCl / 50mM NaOHの混合溶液のところにランニング緩衝液を充填したバイアルに入れ替えてしまえば良いです。もちろんこの方法ですと画面に表示されている溶液とは異なる溶液がセットされていることになりますから、後々見返した時に整合性が取れなくなります。それを避けるためには3回の1M NaCl / 50mM NaOHの混合溶液のコマンドを削除したmethodを作成していただく必要があります。方法は以下の通りです。
Insightの場合
画面右上のEnter custom modeにてNaCl/NaOHのコマンドをRemove
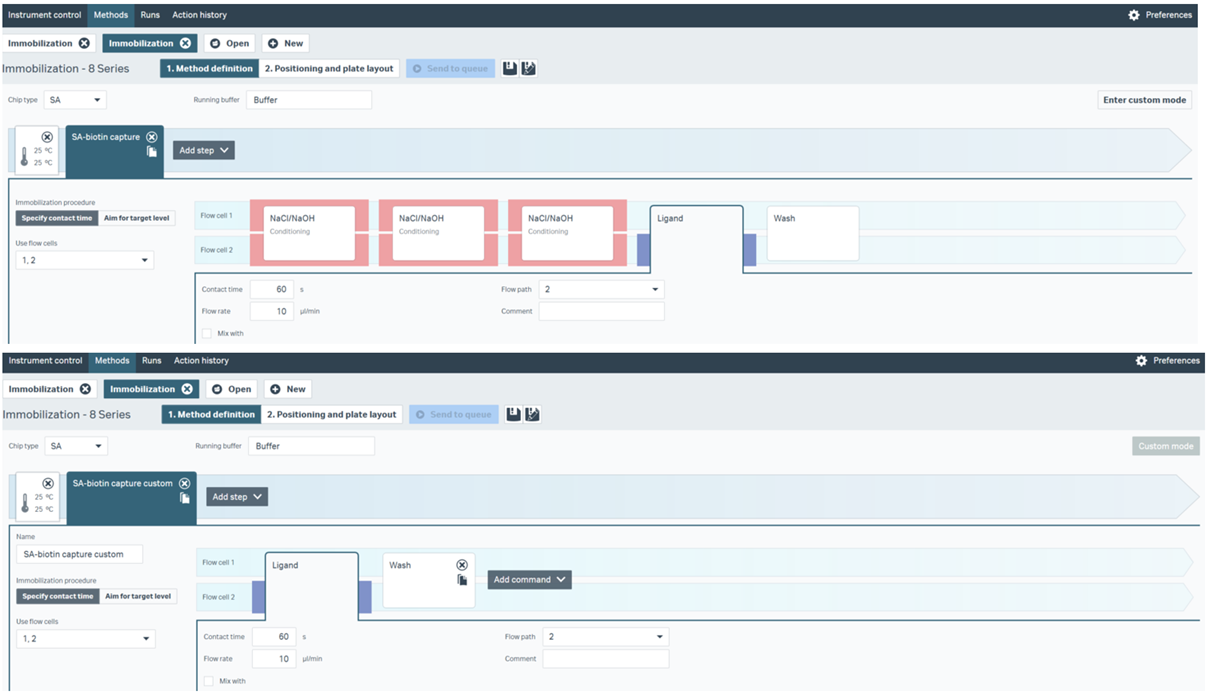
Figure 5 Insightでの編集
Biacore™ T200/S200/X100の場合
Custom Methodを開き、SA-biotin captureを選択>Copyして3回のNaCl/NaOHをDelete>ハイフン(-)がMethod名に入っていると保存できないため、適当な名前に変更してOKで保存>Methodのプルダウンから作成したものに変更
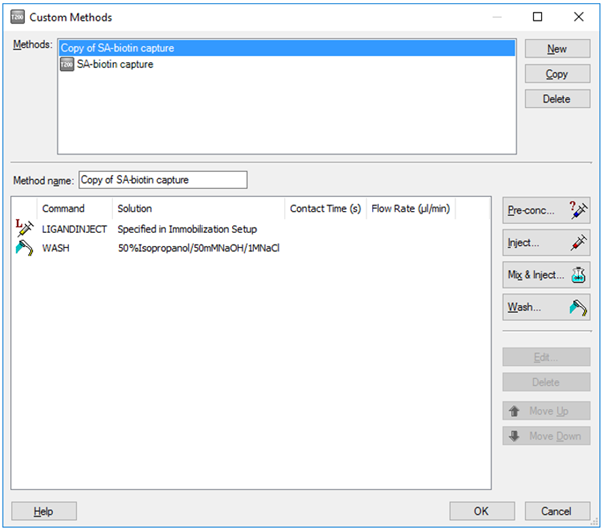
Figure 6 Biacore™ T200/S200/X100での編集
Sensor Chip SAの耐性と再生操作について
本記事の一番下「参考」にもIFUのリンクが貼られていますので詳細はこちらでご確認ください。再生溶液の観点でよく利用される溶液を見てみますと、1分程度のパルスでの添加でしたら、例えば100mM Glycine pH1.5 to 3.0、100mM HCl、300mM Imidazole、4M MgCl2、5M NaCl、100mM NaOH、8M Urea、100% Ethylene glycolなどに耐性があるため、かなり自由に再生溶液を利用することが可能といえます。
参考
Instruction For Useはぜひご確認ください。