「ファージライブラリによるペプチドリガンドのデザインにおける相互作用解析」
鹿児島大学理工学研究科
畠中孝彰 様、伊東祐二 様
はじめに
我々はこれまで、T7ファージディスプレイシステムを利用したライブラリ技術を用いて、標的分子に特異的な結合活性を有するペプチドのデザインに関する研究を行ってきました(1,2)。ここでは、前半に、ヒトIgA (hIgA)を標的として、ファージライブラリを使って得られた特異的な結合ペプチドのBiacore™による親和性解析と、その親和性増強の過程で明らかになった相互作用の熱力学的特性について述べます。後半では、Biacore™を用いてT7ファージ上に提示されたペプチドと標的分子との相互作用を直接解析する手法について紹介します。
なぜヒトIgA?
ヒトIgA(hIgA)は、体内において一日に最も多く生産される抗体のクラス(66 mg/kg/day)であり、バクテリアやウイルスなどに対する最初の防衛ラインである粘膜免疫において主要な役割を担っています(3)。また、近年、血液中IgAは、IgAレセプター(Fcαレセプター/CD89)を介し、免疫細胞中で最も多い好中球をエフェクター細胞として機能させることで、癌細胞などに対する細胞傷害活性(オートファジー)を誘導することが報告されました(4, 5)。これらのことから、hIgAもhIgGと同様に、抗体医薬としての有用性を十分に有していると考えられ、新しい抗体医薬候補として注目されはじめています(6)。しかしながら、IgGにおけるプロテインAのように、産業応用可能なhIgA特異的リガンドが無く、未だhIgAの精製システムが確立されていないというのが現状です。
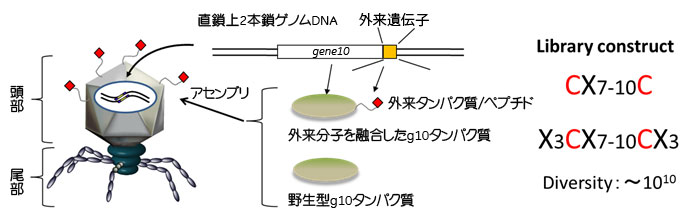
図1 T7ファージディスプレイシステムの概要
頭部を構成するg10タンパクのC末端側に外来のタンパク/ペプチドを提示することができます(7)。抗体(単鎖Fv, Fab)などのタンパク質の提示に従来より多く用いられているM13ファージ(繊維状ファージ)とは異なり、T7ファージはホストである大腸菌から溶菌により放出されるため、膜透過過程を経ないことから提示分子のアミノ酸配列に偏りが少ないという特徴を持ちます(8)。また、大腸菌への感染から溶菌完了までわずか2時間程度で完了するため、特異的な結合ファージを濃縮する手法であるバイオパンニングやその後のスクリーニングにかかる時間を大きく短縮することが出来るといった利点があります。
IgA結合ペプチドのデザイン
我々は、新たなhIgA精製用の特異的アフィニティリガンドをデザインするために、T7ファージディスプレイシステムを利用したランダムペプチドライブラリ(図1)を用いました。図1に示すX3CX7-10CX3型のペプチドライブラリを用いて、IgAに対するバイオパンニングを5ラウンド行った結果、4種類のIgA結合ファージクローンの単離に成功しました(表1)。中でも最もIgAに対する結合力の高かった、A2ファージの提示ペプチドについて合成ペプチドを調製し、IgAに対する親和性を評価した結果、Kd値で1.3 μMと弱い結合力しか示さないことがわかりました(図2)。
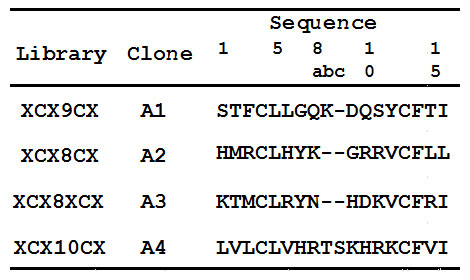
表1 ランダムペプチドライブラリから単離されたIgA結合ペプチド配列
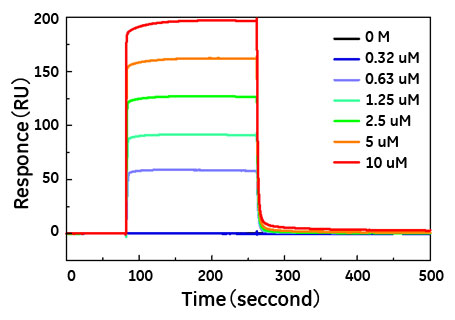
図2 A2ペプチドのIgAに対する結合センサーグラム
CM5センサーチップにhIgAを固定化し、0.32~10 μMのA2ペプチドを流速50 μl/min で180秒間インジェクトし、結合反応を観測した。測定は、HBS-EP緩衝液(pH7)中、25°Cで行った。
そこで、親和性を改善するため、A2ペプチド配列を基に部分ランダムペプチドライブラリを作製し、再度IgAに対するバイオパンニングを行いました。その結果、約30種のペプチドファージが単離され、これらの配列解析の結果、4カ所に完全の保存された結合に必須の残基が同定されましたが、最も親和性の高かったA2-3aファージの提示するペプチドでも、Kd値で530 nMと親和性としては不十分なままでした。
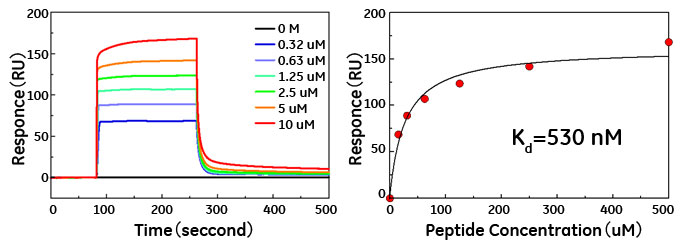
図3 A2-3aペプチドのIgAに対する結合センサーグラム
CM5センサーチップにhIgAを固定化し、0.32~10 μMのA2-3aペプチドを流速50 μl/min で180秒間インジェクトし、結合反応を観測した。測定は、HBS-EP緩衝液(pH7)中、25°Cで行った。
そこで、先の部分ランダムライブラリから得られた配列の中に結合力の向上に関わる配列が含まれていると考え、A2-3aペプチド配列をベースにして、①部分ランダムライブラリ得られた中で出現頻度の高いアミノ酸(表2)への置換体、②各アミノ酸部位のAla置換体、また、③元のA2由来アミノ酸への置換体を化学合成して、hIgAに対する親和性を評価し、実際に結合を上昇させるアミノ酸置換の特定を行いました。
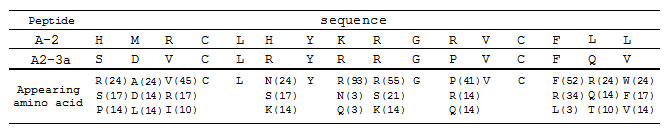
表2 部分ランダムライブラリからの単離されたIgA結合ペプチド配列の各位置における出現アミノ酸と頻度
部分ランダムライブラリより単離された約30種のIgA結合ペプチド配列の各座位において高い出現頻度を示したアミノ酸とその出現頻度(%)を示す。
その結果、表3に示したように、5つの結合力の向上に関わるアミノ酸置換が同定され、さらに、これらの置換を組み合わせたペプチド(Opt-1)を作製したところ、Kd値で33 nMと強い結合を示しました。このOpt-1ペプチドのA2-3aに対する結合力の上昇(1.6 kcal/mol)は、5つの各置換体の結合エネルギーの総和(1.7 kcal/mol)にほぼ相当することから、これらの変異には加成性が成り立ったことが分かります。さらに、Opt-1ペプチドの疎水性を減少させるように、Ala/Ser置換を2カ所導入したOpt-2ペプチド(図4B)は、IgAに対して16 nMのKd値で結合しました。
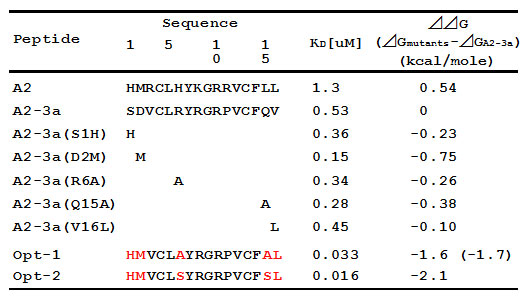
表3 親和性の上昇したA2-3aペプチド変異体
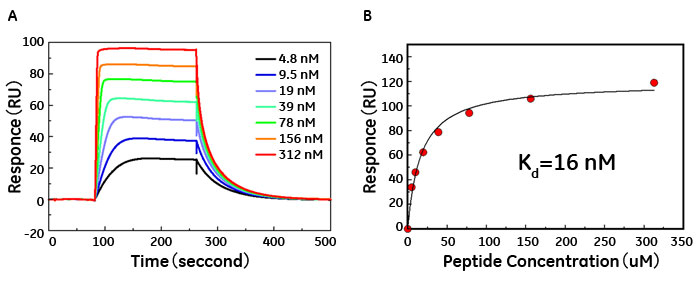
図4 Opt-2ペプチドのIgAに対する結合センサーグラム
CM5センサーチップにhIgAを固定化し、4.8-312 nMのA2-3aペプチドを流速50 μl/minで180秒間インジェクトし、結合反応を観測した。測定は、HBS-EP緩衝液(pH7)中、25°Cで行った。
ペプチドのIgAに対する親和性上昇メカニズム
以上のようにT7ファージディスプレイシステムを用いたライブラリと、合成ペプチドを用いた変異解析により、IgAに対する親和性を1.3 μMから16 nMへと約80倍上昇させることに成功しました。この親和性の向上の機構について考察するために、CDスペクトルによる2次構造分析、Biacore™ T100を用いたIgAとの結合における熱力学的パラメータ解析を行いました。
A2ペプチド、A2-3aペプチド、Opt-2ペプチドのCDスペクトルを図5Aに示しますが、A2ペプチドでは、ほぼランダム構造なのに対し、A2-3a、Opt-2では明らかに、222 nmの負の吸収が大きくなり、αへリックスの2次構造を形成していることが分かります(図5A)。この様に、2次構造の増大と結合力の増大とが相関していることから、IgAとの結合状態のペプチド構造は、αへリックスを有していること示唆されました。ここには示していませんが、Opt-1とIgAのFcとの結合の分子シミュレーション結果から、IgA結合ペプチドはN末端側にヘリックス構造を持った形で結合することが示されており、これらの観察結果を強く支持しています。
次にこれらの3つのペプチドの結合反応を5~35°Cの範囲において測定し、 van't Hoff解析により熱力学的パラメータを得ました。エンタルピー変化(ΔH)については、A2 (-10.9 kcal/mol)とA2-3a (-10.7 kcal/mol) ペプチド間でほぼ変化はなく、Opt-2ペプチド(-11.2 kcal/mol)で、若干(0.3-0.5 kcal/mol)の減少が見られました。一方で、エントロピー項(-TΔS)は 、A2ペプチド(3.1 kcal/mol) に比べ、A2-3aペプチド(1.7 kcal/mol)、Opt-2ペプチド(1.3 kcal/mol)で、それぞれ、1.4、1.8 kcal/molの減少が観察されました。このことは、結合力の向上は、エンタルピーの変化よりもむしろ、エントロピー項の減少によってもたらされていることを示しています。
以上のことから、A2ペプチドからA2-3aさらにはOpt-2への結合力の改善は、アミノ酸置換によるIgAのFcとの直接的な相互作用による結合の増加というよりも、αへリックスを持つ結合構造への平衡的なシフトを促し、ペプチドのチェーンエントロピーを減少させた結果であると考えられます。
このようにペプチドリガンドの親和性の向上においては、分子間の水素結合等による直接の相互作用に関与する残基だけでなく、結合構造をコンフォメーション的に安定化するアミノ酸置換も重要であること、少なくとも本研究のIgA結合ペプチドの場合には、それらの置換は加成性を示すことがわかりました。
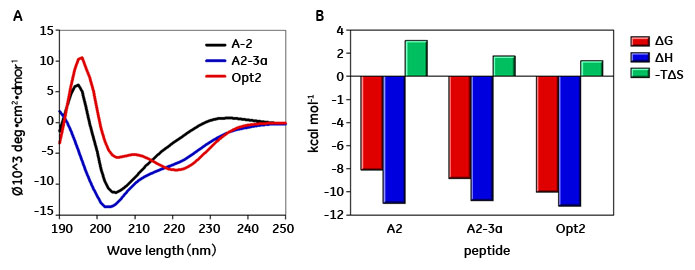
図5 IgA結合ペプチドの二次構造とIgAとの結合における熱力学的パラメータ―
A) A2,A2-3a,Opt-2ペプチドについてリン酸バッファー( Na2HPO4;8.1 mM, KH2PO4 ; 1.5 mM, KCl ; 2.7 mM, NaCl ; 68 mM, pH 7.4)中、J-820(日本分光)を用いて、CDスペクトルを測定した。B) Biacore™ T100を用いて、5~35°CにおけるA2,A2-3a,Opt-2ペプチドとIgAの親和性を解析し、van't Hoffの式 dlnKd=-ΔH/R d(1/T)より、ΔHを算出した。さらに、ギブスエネルギー変化の式ΔG=RTlnKd=ΔH-TΔSより、ΔSを算出した。
T7ファージ上に提示されているペプチドの直接相互作用解析
ファージライブラリによって同定された特異的ペプチドモチーフの正確な親和性解析には、合成ペプチドを準備することが今のところ必須ですが、これはコストも手間もかかるステップです。そこで、我々は、ファージ上に提示されたペプチドの、より正確な親和性の評価について検討を行ってきました。最後に、この解析手法について現状を紹介します。
T7ファージ上に提示されているペプチドの標的分子に対する結合は、ELISAなどの手法により評価することが一般的ですが、Biacore™を使っても簡便に評価することが可能です。図6Aに示したように、標的分子をセンサーチップに固定化し、親和性ペプチドを提示するT7ファージをインジェクトした場合には、結合反応に由来するRUの直線的な増加と極めて遅い解離反応を確認することができます。しかし、この解析では、親和性の差や結合解離反応速度等の評価はできません。図6Aで用いている4-28、5-32、5-50のファージが提示するペプチドの親和性は、本来Kd値で、16,000 nM、360 nM、129 nMとかなり異なりますが、いずれのセンサーグラムも同じような形を取っています。これは、T7ファージ上に複数の分子が提示されているため、この場合のセンサーグラム上に現れる結合反応は、アビディティ効果や再結合の効果により、ペプチドリガンドと標的分子間のアフィニティよりもかなり強いみかけの結合を示してしまうことが原因と考えられます。
そこで我々は、T7ファージを固定化し、ファージ上に提示されたペプチドと標的分子(アナライト)との親和性を直接解析する手法を検討しました。概要としては、CM5センサーチップ上に抗T7ファージ抗体を固定化し、これに、T7ファージを流してキャプチャーさせ、次に濃度が既知の標的分子をインジェクトすることで提示ペプチドと標的分子の親和性を解析するという手法です。固定化するファージ量は、RU値を見ながらコントロールし、Rmaxを200 RU以下に抑えることで、アビディティ効果や再結合効果を最小限に抑えることができます。上記と同じファージをセンサーチップに固定化し、標的分子をインジェクトした場合、ファージ上ペプチドと標的分子間の結合・解離反応を、合成ペプチドの場合と近い形でモニターすることができました(図6B、C)。現在のところ、合成ペプチドと完全に同じ親和性パラメータを得るまでには至っていませんが、ファージ上に提示されたペプチドのある程度正確な結合力の評価、ランキングができるようになっています。
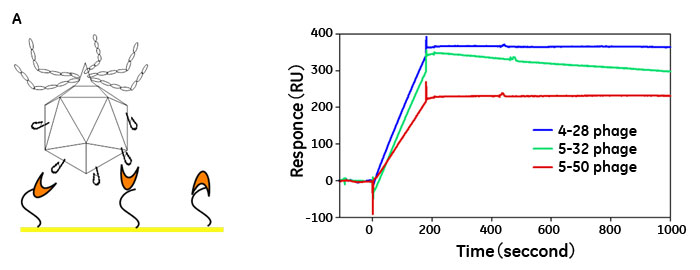
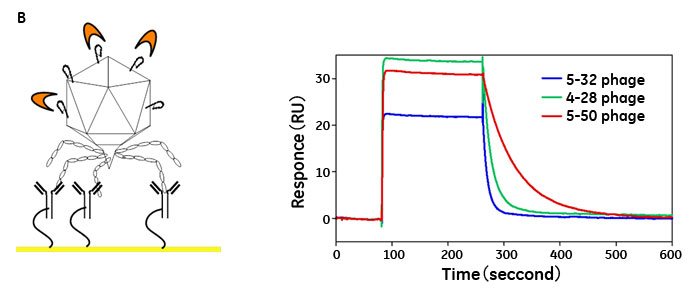
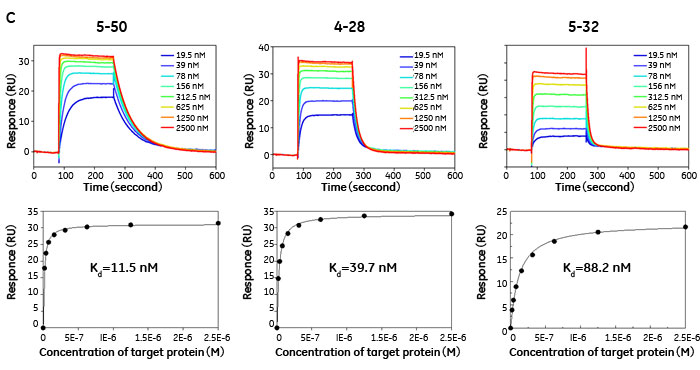
図6 Biacore™を用いたT7ファージ上提示ペプチドの標的に対する親和性解析
A) CM5センサーチップ上に標的分子を固定化し、特異的結合ファージをインジェクトした。B) CM5センサーチップ上に固定化した抗T7ファージ抗体を介してT7ファージをキャプチャーし、標的分子(1 .25μM)をインジェクトした。C) T7ファージを固定化したセルに対して19.5-2,500 nMの標的分子を流速50 μl/min で180秒間インジェクトし、結合反応を観測した。測定は、HBS-EP緩衝液(pH7)中、25℃で行った。解析においては、5-50、4-28、5-32に対する結合レスポンスから、Wild Type ファージへの結合レスポンスを差し引いている。
おわりに
我々は、ファージディスプレイ法を利用したペプチドライブラリを用いて単離したIgA特異的ペプチドの親和性解析を行い、最初に得られたペプチドをベースに親和性向上のストラテジーを組むことによって、高い親和性を有するIgA結合ペプチドのデザインに成功しました。今後、これらの手法とともに、ファージに提示されたペプチドの正確な親和性解析が可能になれば、様々な標的分子に対する迅速かつ低コストのペプチドリガンド探索研究の進展が期待されます。
References
- Sakamoto K, Ito, Y, Mori, T, Sugimura, K. Interaction of human lactoferrin with cell adhesion molecules through RGD motif elucidated by lactoferrin-binding epitopes. The Journal of biological chemistry. 2006; 281: 24472-8.
- Sakamoto K, Ito, Y, Hatanaka, T, Soni, PB, Mori, T, et al. Discovery and characterization of a peptide motif that specifically recognizes a non-native conformation of human IgG induced by acidic pH conditions. The Journal of biological chemistry. 2009; 284: 9986-93.
- Fagarasan S, Honjo, T. Intestinal IgA synthesis: regulation of front-line body defences. Nat Rev Immunol. 2003; 3: 63-72.
- Dechant M, Beyer, T, Schneider-Merck, T, Weisner, W, Peipp, M, et al. Effector mechanisms of recombinant IgA antibodies against epidermal growth factor receptor. J Immunol. 2007; 179: 2936-43.
- Zhao J, Kuroki, M, Shibaguchi, H, Wang, L, Huo, Q, et al. Recombinant human monoclonal igA antibody against CEA to recruit neutrophils to CEA-expressing cells. Oncol Res. 2008; 17: 217-22.
- Beyer T, Lohse, S, Berger, S, Peipp, M, Valerius, T, et al. Serum-free production and purification of chimeric IgA antibodies. Journal of immunological methods. 2009; 346: 26-37.
- Alan Rosenberg KG, F. William Studier, Mark McCormick,James Berg, Robert Novy, and Robert Mierendorf. T7 select phage display system: a powerful new protein display system based on bacteriophage T7. Innovations. 1996; 6: 1-6
- Krumpe LR, Atkinson, AJ, Smythers, GW, Kandel, A, Schumacher, KM, et al. T7 lytic phage-displayed peptide libraries exhibit less sequence bias than M13 filamentous phage-displayed peptide libraries. Proteomics. 2006; 6: 4210-22.












