|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Location:Home > 実験手法別製品・技術情報 > BIA(生物物理学的相互作用解析) > 相互作用解析の王道 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
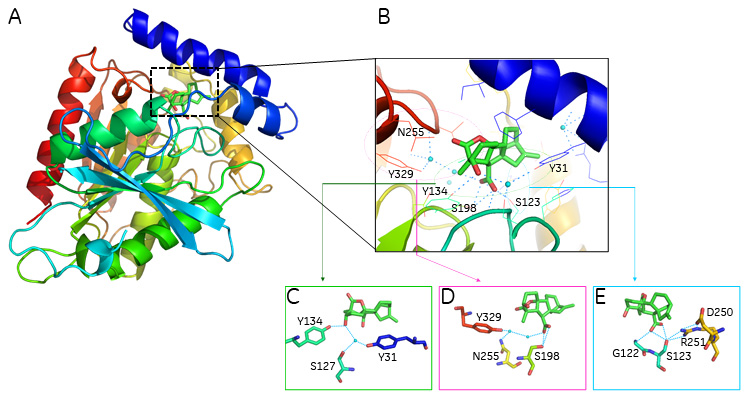
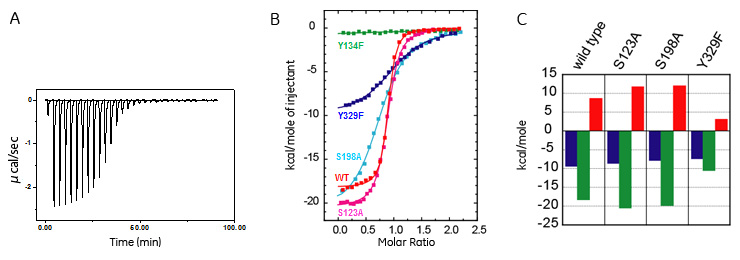
熱分析とタンパク質立体構造に基づくリガンド認識機構の解析(独)農業生物資源研究所 植物科学研究領域 上級研究員 はじめにタンパク質の機能を原子レベルで理解するためには、溶液NMRやX線結晶解析から得られる立体構造情報が欠かせません。タンパク質は生体内において、多くの場合”動き(ダイナミクス)“を伴って機能します。このため、動きを伴った構造(動的構造)情報が得られる溶液NMRによるタンパク質の立体構造解析は、タンパク質の機能を真に理解するために有用な手法として注目されています(例えば筆者らの例として[1])。しかし、溶液NMRには分子量の制限があるために、解析できるタンパク質に限りがあります。一方、結晶構造解析は、結晶化技術の向上や高輝度放射光の充実により、ハイスループットで構造情報が得られる有力な手法です。タンパク質を結晶化させて解析する結晶構造は、タンパク質が取りうる安定構造の一つ(静的構造)を反映していることになります。このため、結晶構造はまれに、結合活性が異なる複合体について同一の構造が得られ、結合活性の違いを結晶構造から説明できない場合があります。このような場合、結晶構造を基盤として、熱力学データを解析することにより、溶液中の分子認識をより深く理解することが可能になると考えられます。ここでは、結晶構造では区別できなかった植物ホルモンの一つであるジベレリン(GA)とその受容体(GID1)の認識機構について、複合体の立体構造解析の結果からだけではわからなかった結合様式の違いを明らかするために、等温滴定型カロリメトリー(ITC)およびコンピューターシミュレーションを併用した解析研究成果[2]について紹介します。 ジベレリンとジベレリン受容体複合体の立体構造を詳細に解析すると、複合体形成には3種類の水素結合ネットワークが重要な因子(図1B)であることが分かりました。GID1のGA認識機構を解明するためにITCによる相互作用解析を行った結果、両者の相互作用は、エンタルピー支配的でした。この結果は、GID1とGAの相互作用が主に水素結合による相互作用であるという結晶構造からの情報とよく一致していました(図2A)。
ITCによるGID1変異体とGAとの相互作用解析GAは植物の伸長や種子の発芽などを促進する植物ホルモンです。GID1は、GAの核内受容体[3]であり、GAを認識後GAシグナル伝達の抑制因子であるSLR1と結合し、SLR1を分解に導くことが明らかとなっています[4]。2008年、イネ(Os)GID1/GA[5]およびシロイヌナズナ(At)GID1/GA/SLR1[6]複合体の立体構造が相次いで報告され、その認識機構の一端が明らかになりました(図1A : OsGID1/GA4複合体構造 pdb id 3EBL)。しかし、活性型GA間の結合活性の違いや、GID1(S123A)やGID1(S198A)変異体とGAとの結合定数の違いについては明確な説明ができませんでした。
さらに我々は、GID1/GAの複合体形成メカニズムを明らかにするために、4種類のGID1変異体(S123A、Y134A、S198A、Y329F)を作成しITC測定を行いました(図2Bおよび表1)。 表1. ITC実験によるOsGID1変異体とGA4の結合定数と熱力学的パラメーター
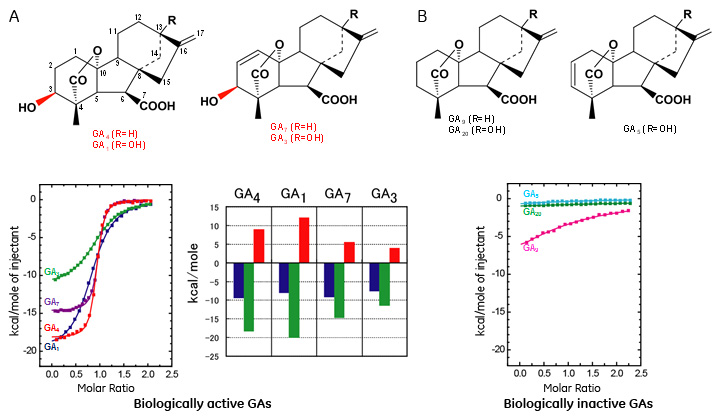
Kb :結合定数、 n:ストイキオメトリー、ΔG:ギブスエネルギー変化、ΔH:エンタルピー変化、-TΔS :エントロピー変化 Y134F変異体は完全にGA4(活性型GAの一つ)との結合活性を失い、Y329F変異体の結合活性は大きく低下しました。この結果は、GA4のC3位の水酸基およびY134とY329の側鎖の水酸基を中心とした水素結合ネットワークが重要であることを示しています。S198A変異体は、Y329と共に形成している重要な水素結合ネットワークが壊れることによりGA4結合活性に大きな影響を及与えました。一方、S123A変異体は、予想に反してGA4結合活性に大きな影響を与えなませんでした。GA4のC6位のカルボキシル基は、S123の側鎖以外にも、G122の主鎖、R251およびD250の側鎖と水素結合しており、大きな影響を及ぼさなかったものと思われます。ここで興味深いのは、水素結合が消失しているにもかかわらずS123A変異体のΔH(-20.5 kcal/mol)の絶対値がwild type(-18.3 kcal/mol)よりも大きいことです。これは、S123の変異によりGA4のC6位のカルボキシル基とR251の側鎖の配向が変わり、両者の間でより安定な水素結合を形成していることが予測されました。これを検証するためにSybyl 3.1.1を用いてS123A変異体のモデリング構造を構築し、Surflex-DockによるとGA4のドッキングシミュレーションを行いました。その結果、(結晶構造およびリドッキングシミュレーション構造とも)wild typeではGA4のC6位のカルボキシル基とR251の側鎖間の水素結合距離が3.8-4.1 Åでしたが、S123A変異体では2.9-3.2 Åと短くなっており、ITCを用いた実験結果をサポートしていました。 ITCによるGID1と各種GAとの相互作用解析植物体内にはGAが130種類以上存在することが知られています。その中で活性型として存在するのは4種類だけです。これらGAについて正確な結合活性と熱力学的活性データを得るためにITC測定を行いました(図3)。
その結果、非活性型GAはGA9を除いてGID1と全く結合しませんでした(図3B)。このことから、立体構造からも予想されたように、活性型GAに存在する(前章で着目したY134と直接水素結合をしている)3位の水酸基がGID1との結合に重要であることが分かりました。活性型GAは、それぞれ熱力学的寄与が大きく異なることが分かりました(図3A)。例えば、GA4とGA7、GA1とGA3はそれぞれ結合定数が同じオーダーであるにも関わらず、GA4とGA1は、GA7とGA3に比べて、エンタルピー支配的にGID1に結合することがわかりました。 表2. ITC実験によるOsGID1と各種GAの結合定数と熱力学的パラメーター
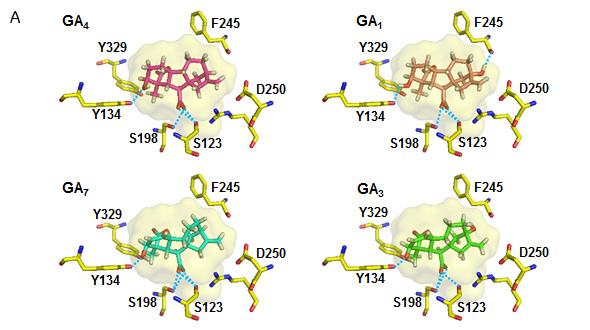
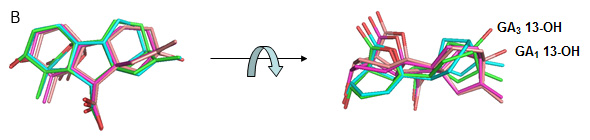
Kb :結合定数、 n:ストイキオメトリー、ΔG:ギブスエネルギー変化、ΔH:エンタルピー変化、-TΔS :エントロピー変化 これらのGAの間には、1位と2位の炭素結合が単結合であるか二重結合であるという違いがあり、そのためGA7とGA3では構造の柔軟性が失われ、水素結合形成が不利になっていること、また二重結合により拘束されているためエントロピー変化に影響を及ぼさないことが予想されました。そこで、これらの結果をドッキングシミュレーションにより検証を行いました。その結果、GA4、GA1とGA7、GA3との間には骨格構造に大きな違いが見られました(図4)。
この結果は、OsGID1/GA4および OsGID1/GA3の立体構造では見られませんでしたが、AtGID1/GA/SLR1複合体におけるGA4とGA3の構造の違いと類似していました。 おわりに溶液状態で測定するITCは、タンパク質結合活性の熱力学的理解に非常に有効です。我々は、GID1のGA認識機構について、ITCによる解析結果を用いて、静的構造である結晶構造情報を補完するより詳細な解析を行うことができました。近年、我々の研究以外にも結晶構造情報と共にITCによる熱力学データが議論されている論文を多く見かけるようになりました。このことからも、結晶構造と熱力学的データの相互利用が有用であることがわかります。今後も、立体構造情報と熱力学的を併用することにより、より深いタンパク質機能の解明が期待されます。 Reference
「相互作用解析の王道」について「相互作用解析の王道」は、2009年8月よりバイオダイレクトメールでお届けしています。
バイオダイレクトメールは弊社WEB会員向けメールマガジンです。バイオダイレクトメールの配信をご希望の方は、下記リンク先からご登録をお願いいたします。 関連リンク津本先生の研究内容や論文などはこちら→津本浩平先生の研究室Webサイト お問合せフォーム※日本ポールの他事業部取扱い製品(例: 食品・飲料、半導体、化学/石油/ガス )はこちらより各事業部へお問い合わせください。 お問い合わせありがとうございます。 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
© 2026 Cytiva